Avaliação do dano a haste capilar ocasionado por tintura oxidativa ...
fosforolização oxidativa
-
Upload
brunowarley1982 -
Category
Documents
-
view
124 -
download
0
Transcript of fosforolização oxidativa
Fosforilao oxidativa
1
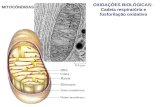
Fosforilao oxidativaA fosforilao oxidativa uma via metablica que utiliza energia libertada pela oxidao de nutrientes de forma a produzir trifosfato de adenosina (ATP). O processo refere-se fosforilao do ADP em ATP, utilizando para isso a energia libertada nas reaces de oxidao-reduo. Durante a fosforilao oxidativa, existe transferncia de eltrons de doadores electrnicos (molculas redutoras) a aceitadores electrnicos (molculas oxidantes), tais como o dioxignio, numa reao de oxido-reduo. As transferncias de eletrns constituem estas reaes de oxido-reduo, que se processam com libertao de energia, biologicamente aproveitvel para a A cadeia de transporte electrnico na mitocndria o local onde ocorre a fosforilao biossntese de ATP. Em eucariontes, oxidativa em eucariontes. O NADH e succinato produzidos no ciclo dos cidos tais reaes redox so feitas por cinco tricarboxlicos so oxidados, libertando-se energia utilizvel pela ATP sintase. complexos principais de protenas mitocondriais, enquanto que em procariontes, diferentes protenas localizam-se na membrana interna da clula, dependendo o tipo de enzima utilizado dos aceitadores e doadores electrnicos. Ao conjunto de complexos proteicos envolvidos nestas reaes chama-se cadeia de transporte. A energia derivada do transporte de eltrons convertida numa fora motriz protenica e principalmente utilizada para bombear prtons para o exterior da matriz mitocondrial. Este processo denominado quimiosmose e origina energia potencial sob a forma de um gradiente de pH (ou seja, uma concentrao diferente de prtons dentro e fora da mitocndria) e de potencial eltrico atravs da membrana. A energia utilizada ao permitir-se o fluxo de prtons a favor do gradiente de concentrao atravs da enzima ATP sintase. Embora a fosforilao oxidativa seja uma parte vital do metabolismo, produz espcies reativas de oxignio tais como o superxido e o perxido de hidrognio, que induzem a propagao de radicais livres, danificando componentes celulares (por exemplo, oxidando protenas e lpidios de membrana) e contribuindo para processos de envelhecimento celular e patologias. Existem tambm diversos venenos e medicamentos que tm como alvo as enzimas desta via metablica, inibindo a sua atividade.
HistriaO campo de estudo da fosforilao oxidativa iniciou-se em 1906, com a divulgao por Arthur Harden de um papel vital do fosfato na fermentao celular, embora fossem conhecidos apenas ento fosfatos de acares.[1] A ligao entre a oxidao de acares e a sntese de ATP foi firmemente estabelecida no incio da dcada de 1940 do sculo XX por Herman Kalckar,[2] confirmando-se o papel central do ATP na transferncia de energia proposto por Fritz Albert Lipmann em 1941.[3] Mais tarde, em 1949, Morris Friedkin e Albert L. Lehninger provaram que a coenzima NADH ligava vias metablicas tais como o ciclo do cido ctrico e a sntese de ATP.[4] Durante as duas dcadas seguintes permaneceu incgnito o mecanismo de produo do ATP, tendo havido a procura de um elusivo
Fosforilao oxidativa "intermedirio de alta energia" que ligaria a oxidao s reaces de fosforilao.[5] Este problema foi resolvido por Peter D. Mitchell com a publicao da teoria quimiosmtica em 1961.[6] A proposta foi inicialmente controversa, mas foi lentamente aceite e Mitchell recebeu um prmio Nobel pelos seus estudos em 1978.[7] [8] A investigao que se seguiu neste campo concentrou-se na purificao e caracterizao das enzimas desta via, havendo contribuies importantes por David E. Green nos complexos da cadeia de transporte electrnico e Efraim Racker na ATP sintase.[9] Importantes passos em direco descoberta do mecanismo da ATP sintase foram dados por Paul D. Boyer com a sua proposta do mecanismo "ligao-modificao" em 1973 e de catlise envolvendo rotao em 1982.[10] [11] O trabalho mais recente no campo da fosforilao oxidativa inclui estudos estruturais das enzimas desta via por John E. Walker (tambm conhecido como Johnnie Walker) tendo Walker e Boyer recebido um prmio Nobel em 1997.[12]
2
A transferncia de energia pela quimiosmoseEmbora as diversas formas de vida na Terra utilizem uma larga gama de nutrientes diferentes, quase todas usam a fosforilao oxidativa para produo de ATP, a molcula que fornece energia metablica. Esta via to universal provavelmente por ser uma forma altamente eficiente de armazenar energia, comparando com processos alternativos de fermentao como a gliclise. A fosforilao oxidativa funciona utilizando reaces qumicas exergnicas para dar energia a reaces endergnicas; os dois tipos de reaco dizem-se, neste caso, acoplados, ou seja, um no ocorre sem o outro. O fluxo de electres atravs da cadeia de transporte electrnico, desde doadores electrnicos como o NADH a aceitadores de electres como o oxignio, um processo exergnico, ou seja, liberta energia, enquanto que a sntese da ATP endergnica e requer portanto energia. A variao de energia livre associada transferncia de electres atravs de um dos trs complexos corresponde a uma fora motriz protnica capaz de fazer a sntese de ATP. Para cada NADH que se oxida, ou seja, para cada par de electres transportado pelos complexos I, III e IV, h a sntese de trs ATP. Tanto a cadeia de transporte electrnico como a ATP sintase se localizam numa membrana. A energia transferida da cadeia de transporte electrnico para a ATP sintase pelo movimento de protes atravs da membrana, num processo denominado quimiosmose.[13] Como a membrana interna da mitocndria impermevel a protes, estes s podem voltar matriz e desfazer o gradiente atravs de stios especficos da membrana interna. O transporte de protes atravs desta feita pelos complexos I, III e IV. A ATP sintase utiliza a energia para sintetizar ATP a partir da fosforilao de difosfato de adenosina (ADP). Parte da enzima sofre uma rotao medida que os protes passam por ela. Na prtica, este processo semelhante a um simples circuito elctrico, existindo uma corrente de protes do lado negativo (N) da membrana para o lado positivo (P) provocada pela aco de enzimas da cadeia de transporte electrnico que bombeiam esses protes. Estas enzimas actuam como uma pilha, medida que produzem trabalho circulando corrente atravs do circuito. O movimento de protes cria um gradiente electroqumico atravs da membrana, muitas vezes designada como fora motriz protnica. Este gradiente tem duas componentes: uma diferena na concentrao de protes (gradiente de pH) e uma diferena no potencial elctrico, tendo o lado N uma carga negativa. A energia armazenada principalmente sob a forma de uma diferena de potenciais elctricos nas mitocndrias e sob a forma de gradiente de pH nos cloroplastos.[14] A ATP sintase liberta esta energia armazenada ao completar o circuito e permitir o fluxo de protes ao longo do potencial electroqumico, de volta ao lado N da membrana.[15] Esta enzima actua como um motor elctrico, ao usar a fora motriz protnica para fornecer energia rotao de parte da sua estrutura e acoplar este movimento sntese de ATP. A quantidade de energia libertada pela fosforilao oxidativa alta, comparando-se com a quantidade de energia produzida pela fermentao anaerbia. A gliclise produz apenas duas molculas de ATP, enquanto que a fosforilao oxidativa produz, a partir de dez molculas de NADH e duas de succinato, 26 molculas de ATP, comparando-se a converso de uma molcula de glicose a dixido de carbono e gua.[16] Este rendimento de ATP
Fosforilao oxidativa o valor mximo terico; na prtica, alguns protes passam tambm atravs da membrana, baixando o rendimento de produo de ATP.[17]
3
Molculas de transferncia de protes e electresA cadeia de transporte electrnico transporta protes e electres, mediando a passagem de electres de doadores reduzidos a aceitadores electrnicos e transportando protes atravs da membrana. Estes processos tanto usam molculas solveis como grupos ligados a protenas. Nas mitocndrias, os electres so transferidos dentro do espao intermembranar pela protena de transporte electrnico citocromo c,[18] que, por ser hidrossolvel, pode circular no espao intermembranar. O citocromo c transporta apenas electres, atravs da oxirreduo de um io de ferro localizado num grupo hemo pertencente estrutura da protena. Tambm se encontra citocromo c nalgumas bactrias, localizando-se no espao periplasmtico.[19]
Reduo da coenzima Q a partir da sua forma de ubiquinona (Q, cima) forma totalmente reduzida ubiquinol (QH2, em baixo).
Na membrana mitocondrial interna, a coenzima Q10 (Q), um transportador electrnico lipossolvel, transporta no s electres mas tambm protes, usando um ciclo redox.[20] Esta pequena molcula de benzoquinona muito hidrofbica, podendo por isso difundir-se facilmente pela membrana. Quando Q aceita dois electres e dois protes, passa forma totalmente reduzida ubiquinol (QH2); quando QH2 liberta dois protes e dois elecres, volta ao estado ubiquinona (Q). Como resultado, se duas enzimas esto dispostas de modo que Q seja reduzido de um lado da membrana e QH2 seja oxidado no outro lado, a ubiquinona acoplar estas reaces e transportar protes atravs da membrana.[21] Algumas cadeias de transporte electrnico bacterianas usam quinonas diferentes, tais como a menaquinona (ou vitamina K), alm da ubiquinona.[22] Dentro de protenas, os electres so transferidos entre cofactores flavnicos,[15] [23] centros de ferro-enxofre e citocromos. Existem diversos tipos de centros ferro-enxofre. O tipo mais simples que se encontra na cadeia de transporte electrnico formado por dois tomos de ferro ligados entre si e por dois tomos de enxofre inorgnico (ou seja, no pertencente a cadeias laterais de aminocidos), designando-se este tipo de centros [2Fe-2S]. O segundo tipo de centro ferro-enxofre o [4Fe-4S], sendo similar a um cubo constitudo por quatro ies de ferro e quatro de enxofre. Nos centros de ferro-enxofre, cada io de ferro encontra-se coordenado tambm a um aminocido, normalmente atravs do tomo de enxofre de uma cistena. Os cofactores contendo metais sofrem reaces redox sem ligar ou libertar protes, pelo que servem apenas para transportar electres na cadeia de transporte electrnico. Os electres conseguem viajar distncias relativamente grandes dentro das protenas ao efectuar "saltos" entre as cadeias dos cofactores.[24] Tal ocorre devido ao efeito de tunneling quntico, que rpido atravs de distncias inferiores a 14.[25]
Cadeias de transporte electrnico em eucariontesDiversos processos bioqumicos catablicos, tais como a gliclise, o ciclo dos cidos tricarboxlicos e a beta-oxidao, produzem a coenzima NADH. Esta coenzima contm electres que possuem um alto potencial de transferncia (correspondente a um potencial de elctrodo muito negativo), ou seja, ao acontecer a oxidao do NADH, libertada grande quantidade de energia. No entanto, a clula no liberta esta energia de uma s vez, pois tal reaco poderia ser incontrolvel. Os electres so ento removidos do NADH e transferidos para o dioxignio atravs de uma srie de passos catalisados por diferentes enzimas, em que cada passo liberta uma pequena
Fosforilao oxidativa quantidade de energia. Este conjunto de enzimas, designados complexos I, II, III e IV, constitui a cadeia de transporte electrnico e encontra-se na membrana interna da mitocndria. O succinato tambm oxidado pela cadeia de transporte electrnico, mas entra na via metablica num ponto diferente. Em eucariontes, as enzimas neste sistema de transporte electrnico utilizam a energia libertada na oxidao do NADH para bombear protes atravs da membrana interna da mitocndria. Esta aco causa a acumulao de protes no espao intermembranar, originando um gradiente electroqumico atravs da membrana. A energia armazenada sob este potencial ento utilizada pela ATP sintase para produzir ATP. A fosforilao oxidativa mitocondrial a mais bem compreendida; existem mitocndrias em quase todos os eucariontes, exceptuando-se alguns protozorios anaerbios como Trichomonas vaginalis, que reduzem os protes a hidrognio molecular num organelo denominado hidrogenossoma, uma mitocndria residual.[26]
4
NADH-coenzima Q oxidoredutase (complexo I)A NADH-coenzima Q oxidorredutase, tambm conhecida como NADH desidrogenase ou complexo I, a primeira protena na cadeia de transporte electrnico.[27] O complexo I uma enzima de grandes dimenses; o complexo I de mamferos possui 46 subunidades e uma massa molecular de cerca de mil quilodaltons.[28] conhecida apenas a estrutura detalhada do complexo de uma bactria;[29] na maioria dos organismos, o complexo aparenta ter a forma de uma bota com uma esfera projectando-se da membrana em direco matriz mitocondrial.[30] [31] Os genes que codificam as protenas que fazem parte deste complexo encontram-se tanto no DNA nuclear como no genoma mitocondrial, tal como acontece com diversas outras enzimas presentes na mitocndria.
Complexo I ou NADH-Q oxidorredutase. As abreviaturas utilizadas encontram-se discutidas no texto. Em todos os diagramas de complexos respiratrios, a matriz mitocondrial situa-se em baixo e o espao intermembranar em cima.
A reaco catalisada por esta enzima a reduo da coenzima Q10 (ou ubiquinona, representado por Q na equao abaixo) por dois electres provindos do NADH. A coenzima Q10 uma quinona lipossolvel da membrana mitocondrial.
O incio da reao, e de toda a cadeia electrnica, consiste na ligao de uma molcula de NADH ao complexo I e a doao de dois electres. Os electres entram no complexo I atravs de um grupo prosttico ligado ao complexo, o mononucletido de flavina (FMN). A adio de electres ao FMN converte este sua forma reduzida, FMNH2. Os electres so ento transferidos atravs de diversos centros de ferro-enxofre, o segundo tipo de grupo prosttico encontrado no complexo.[29] Existem centros [2Fe-2S] e [4Fe-4S] no complexo I. medida que os electres passam atravs deste complexo, quatro protes so bombeados da matriz mitocondrial para o espao intermembranar. No bem conhecido o mecanismo exacto de como esta passagem ocorre, mas
Fosforilao oxidativa aparenta haver mudanas conformacionais no complexo I que provocam a ligao de protes ao lado N da membrana e os movimentam para o lado P.[32] Por fim, os electres so transferidos da cadeia de centros ferro-enxofre para uma molcula de ubiquinona na membrana.[27] A reduo da ubiquinona contribi tambm para a gerao de um gradiente de protes, por haver retirada de dois protes da matriz na sua reduo a ubiquinol (QH2).Este processo se tornou importante, pois, seres heterotrficos necessitam deste ciclo(terceira fase do processo de transformao quimica da glicose).
5
Succinato-Q oxidorredutase (complexo II)A succinato-Q oxidorredutase, tambm conhecida como complexo II, um segundo ponto de entrada na cadeia de transporte electrnico.[33] Tem a caracterstica de ser a nica enzima que participa tanto no ciclo dos cidos tricarboxlicos como na cadeia de transporte electrnico. O complexo II consiste de quatro subunidades proteicas e um cofactor dinucletido de flavina-adenina (FAD), centros de ferro-enxofre e um grupo hemo que no participa na transferncia de electres para a coenzima Q mas aparenta ser necessrio para diminuir a produo de espcies reactivas de oxignio.[34] [35] Oxida o succinato a fumarato e reduz a ubiquinona. Como esta reaco liberta menos energia que a oxidao do NADH, o complexo II no transporta protes atravs da membrana e no contribui para o gradiente de protes.
Complexo II: Succinato-Q oxidorredutase.
Nalguns eucariontes, tais como o verme parasita Ascaris suum, existe uma enzima similar ao complexo II, a fumarato redutase (menaquiol:fumarato oxidorredutase, ou QFR) que opera de forma reversa, oxidando ubiquinol e reduzindo fumarato. Este processo permite ao parasita sobreviver no ambiente anaerbio do intestino grosso, realizando fosforilao oxidativa anaerbia usando fumarato como aceitador final de electres.[36] Outra funo pouco convencional do complexo II encontrada no parasita que causa a malria Plasmodium falciparum, em que a aco reversa do complexo II importante na regenerao de ubiquinol, utilizado pelo parasita num tipo raro de biossntese de pirimidina.[37]
Flavoprotena de transporte de electres-Q oxidorredutaseA flavoprotena de transporte de electres-ubiquinona oxidorredutase (ETF-Q oxidorredutase), tambm conhecida como flavoprotena de transporte de electres desidrogenase, um terceiro ponto de entrada na cadeia de transporte electrnico. uma enzima que aceita electres da flavoprotena transportadora de electres na matriz mitocondrial e os utiliza para reduzir a ubiquinona.[38] Esta enzima contm uma flavina e um centro [4Fe-4S] mas, ao contrrio de outros complexos respiratrios, liga-se superfcie da membrana e no atravessa a bicamada lipdica.[39]
Em mamferos, esta via metablica relevante na beta-oxidao de cidos gordos e no catabolismo de aminocidos e colina, ao aceitar electres de diversas acetil-CoA desidrogenases.[40] [41] Em plantas, a ETF-Q oxidorredutase tambm importante nas respostas metablicas que permitem a sobevivncia durante longos perodos de escurido.[42]
Fosforilao oxidativa
6
Q-citocromo c oxidorredutase (complexo III)A Q-citocromo c oxidorredutase tambm conhecida simplesmente como citocromo c redutase, complexo citocromo bc1, ou simplesmente complexo III.[43] [44] Em mamferos, esta enzima um dmero, em que cada subunidade ela prpria um complexo de 11 protenas, um centro [2Fe-2S] e trs citocromos (um citocromo c1 e Os dois passos de transferncia electrnica no complexo III: Q-citocromo c oxidorredutase. Aps cada passo, Q (na parte superior da figura) deixa a enzima. dois citocromos b.[45] Um citocromo um tipo de protena de transferncia electrnica que contm pelo menos um grupo hemo. Os ies de ferro dos grupos hmicos do complexo III alternam entre o estado ferroso (reduzido, Fe2+) e frrico (oxidado, Fe3+), medida que os electres so transferidos atravs da protena. O complexo III catalisa a oxidao de uma molcula de ubiquinol e a reduo de duas molculas de citocromo c, que consegue transportar apenas um electro (ao contrrio da coenzima Q, que pode transportar dois)
Como apenas um dos electres pode ser transferido em cada passo do doador QH2 para um citocromo aceitador, o mecanismo de reaco do complexo III mais elaborado que aqueles de outros complexos respiratrios e ocorre em dois passos colectivamente designados "ciclo Q".[46] No primeiro passo, a enzima liga trs substratos: primeiro o QH2, que sofre oxidao, passando um electro para o segundo substrato, o citocromo c, e dois protes para o espao intermembranar. O terceiro substrato Q, que aceita o segundo electro de QH2, reduzindo-se ao radical Q.(ubisemiquinona). Os primeiros dois substratos so libertados, enquanto que o intermedirio ubisemiquinona permanece ligado. No segundo passo, liga-se uma segunda molcula de QH2, passando novamente um electro a outro citocromo c. O segundo electro transferido para a ubisemiquinona, reduzindo-a a QH2 ao mesmo tempo que so captados dois protes da matriz mitocondrial. QH2 ento libertado da enzima.[47] medida que a coenzima Q reduzida a ubiquinol no lado interno da membrana e oxidada a ubiquinona no outro lado, existe uma transferncia lquida de protes atravs da membrana, que contribui para o gradiente de protes.[15] Este mecanismo relativamente complexo mas assegura um aumento da eficincia da transferncia de protes: se apenas uma molcula de QH2 fosse utilizada para reduzir directamente dois citocromos, a eficincia seria a metade, havendo apenas a transferncia de um proto por citocromo reduzido.[15]
Fosforilao oxidativa
7
Citocromo c oxidase (complexo IV)A citocromo c oxidase, tambm conhecda como complexo IV, o ltimo complexo proteico da cadeia de transporte de electres.[48] Em mamferos, a enzima tem uma estrutura bastante complexa, contendo 13 subunidades, dois grupos hmicos e diversos outros cofactores metlicos (trs ies de cobre, um de magnsio e um de zinco).[49] Esta enzima catalisa a reaco final da cadeia de transporte electrnico, oxidando o citocromo c e transferindo electres para o oxignio, ao mesmo tempo que bombeia protes [50] atravs da membrana. O aceitador final de electres oxignio reduzido a gua neste processo. Tanto a passagem de protes atravs da membrana como o consumo de protes na matriz mitocondrial contribuem para o gradiente protnico.Complexo IV: citocromo c oxidase.
Redutases e oxidases alternativasMuitos organismos eucariticos possuem cadeias respiratrias diferentes das de mamferos, que so as mais bem estudadas (e acima descritas). Por exemplo, em plantas, existem NADH oxidases que oxidam o NADH no citoplasma, no na matriz mitocondrial, e passam os electres para uma reserva de ubiquinona.[51] Estas enzimas no transportam proes, pelo que reduzem a ubiquinona sem alterar o gradiente electroqumico atravs da membrana interna.[52] Outro exemplo de um sistema diferente a "oxidase alternativa", encontrada em plantas, alguns fungos, protistas e possivelmente noutros animais.[53] [54] Esta enzima transfere electres directamente do ubiquinol para o oxignio.[55] As vias de transporte electrnico em que participam estas oxidases alternativas rendem menos ATP que a cadeia completa. No se encontram totalmente esclarecidas as vantagens em possuir cadeias mais curtas; no entanto, estas oxidases alternativas so produzidas em resposta a situaes de stress, como frio, produo de espcies reactivas de oxignio e infeco, assim como outros factores que inibam a cadeia de transporte completa.[56] [57] Vias alternativas podem melhorar a resistncia dos organismos a danos causados pelo stress oxidativo.[58]
Organizao de complexosO modelo original da organizao dos complexos da cadeia respiratria descrevia a sua difuso livre e independente na membrana mitocondrial.[28] No entanto, alguns dados mais recentes sugerem que os complexos possam formar estruturas de ordem superior, designadas "supercomplexos" ou "respirassomas".[59] Neste modelo, os diversos complexos existem como conjuntos organizados de enzimas que interactuam.[60] Tais associaes podero permitir a canalizao de substratos ("channeling") entre os diferentes complexos da cadeia, optimizando a velocidade e eficincia da transferncia de electres.[61] Em mamferos, alguns dos componentes podero existir em maior quantidade que outros, com razes entre complexos I/II/III/IV e ATP sintase de aproximadamente 1:1:3:7:4.[62] No entanto, este modelo no totalmente aceite, pois existem dados que aparentam no se ajustar ao modelo.[28] [63]
Fosforilao oxidativa
8
Cadeias de transporte electrnico de procariontesEm contraste com a similaridade geral que existe na estrutura e funo das cadeias respiratrias em eucariontes, as enzimas de transferncia electrnica em bactrias e arqueas so muito diversificadas; utilizam tambm diversos outros compostos qumicos como substratos, permitindo a sua adaptao a diferentes condies ambientais.[64] [65] Tal como acontece nos eucariontes, a cadeia de transporte electrnico em procariontes utiliza a energia libertada da oxidao de um substrato para bombear ies atravs de uma membrana e gerar um gradiente electroqumico. Em bactrias, a fosforilao oxidativa em Escherichia coli a mais bem compreendida; em contraste, os sistemas em arqueas so ainda pouco compreendidos.[66] Em E. coli, a fosforilao oxidativa utiliza uma grande variedade de agentes redutores e oxidantes, listados abaixo. O potencial de meia onda de um composto d uma medida da quantidade de energia libertada quando esse composto oxidado ou reduzido, tendo agentes redutores potenciais negativos e agentes oxidantes potenciais positivos.Enzimas e substratos da respirao em E. coli Enzima respiratria Formato desidrogenase Hidrogenase NADH desidrogenase Glicerol-3-fosfato desidrogenase Piruvato oxidase Lactato desidrogenase D-aminocido desidrogenase Glicose oxidase Succinato desidrogenase Ubiquinol oxidase Nitrato redutase Nitrito redutase Dimetilsulfxido redutase N-xido de trimetilamina redutase Fumarato redutase Par redox Bicarbonato / Formato Proto / Hidrognio NAD+ / NADH DHAP / Gly-3-P Acetato + Dixido de carbono / Piruvato Piruvato / Lactato 2-oxocido + amnia / D-aminocido Glicose / Gluconato Succinato / Fumarato Oxignio / gua Nitrato / Nitrito Nitrito / Amnia DMSO / DMS TMAO / TMA Fumarato / Succinato [67] Potencial de meia onda (Volts) 0,43 0,42 0,32 0,19 ? 0,19 ? 0,14 +0,03 +0,82 +0,42 +0,36 +0,16 +0,13 +0,03
Como mostrado acima, a E. coli pode multiplicar-se na presena de agentes redutores como o formato, o hidrognio ou o lactato como doadores de electres e o nitrato, DMSO ou oxignio como aceitadores.[65] Quanto maior a diferena entre o potencial de um composto oxidante e de um redutor, mais energia libertada quando eles reagem. Dentro deste conjunto de compostos, o par succinato/fumarato particular, pois o seu potencial de meia onda quase zero. Tal significa que o succinato pode ser oxidado a fumarato se houver um oxidante forte presente (como o oxignio) ou o fumarato pode ser reduzido a succinato na presena de um agente redutor forte (como o formato). Estas reaces alternativas so catalisadas pela succinato desidrogenase e pela fumarato redutase, respectivamente.[68] Alguns procariontes utilizam pares redox que possuem diferenas muito pequenas no seu potencial de meia onda. Por exemplo, bactrias nitrificantes, como as pertencentes ao gnero Nitrobacter, oxidam nitrito a nitrato, doando elecres ao oxignio. A pequena quantidade de energia libertada nesta reaco suficiente para bombear protes e produzir ATP, mas insuficiente para produzir NADH ou NADPH directamente em anabolismo.[69] Este problema contornado usando uma nitrito oxidorredutase que produz fora motriz protnica suficiente para fazer funcionar a cadeia de transporte electrnico no sentido inverso, forando o complexo I a produzir NADH.[70] [71]
Fosforilao oxidativa Os procariontes controlam o uso destes doadores e aceitadores de electres variando o tipo de enzimas produzido, em resposta a condies ambientais.[72] Esta flexibilidade deve-se possibilidade de diferentes oxidases e redutases utilizarem a mesma reserva de ubiquinona. Tal permite diversas combinaes funcionais de enzimas, enzimas essas ligadas pelo intermedirio comum ubiquinol.[67] Estas cadeias respiratrias tm portanto uma natureza modular, com sistemas de enzimas fceis de permutar. Alm da existncia desta diversidade metablica, os procariontes tm tambm vrias isozimas (diferentes enzimas que catalisam a mesma reaco). Por exemplo, existe em E. coli dois tipos diferentes de ubiquinol oxidase usando oxignio como aceitador electrnico. Sob condies totalmente aerbias, a clula utiliza uma oxidase com baixa afinidade para com o oxignio que consegue transportar dois protes por cada electro. No entanto, se os nveis de oxignio decrescem, o metabolismo muda para a utlizao de uma oxidase que transfere apenas um proto por electro, mas que tem alta afinidade para com o oxignio.[73]
9
ATP sintaseA ATP sintase, tambm designada complexo V, a enzima final na via da fosforilao oxidativa. Esta enzima encontra-se presente em todas os organismos vivos e funciona de forma idntica em procariontes e eucariontes. [74] A enzima utiliza a energia armazenada num gradiente de protes existente atravs da membrana para realizar a sntese de ATP a partir de ADP e fosfato inorgnico (Pi). Existem estimativas de serem necessrios entre trs e quatro protes para sintetizar um ATP,[75] [76] havendo alguns estudos que apontam para uma variao nestes nmeros, dependendo das condies.[77]
ATP sintase. O canal de protes FO e eixo encontra-se a rosa, o domnio sintase F1 a magenta e a membrana a azul translcido.
Esta reaco de fosforilao um equilbrio qumico, que pode ser deslocado alterando-se a fora motriz protnica. Se no existe uma fora motriz, a reaco da ATP sintase prossegue da direita para a esquerda, havendo a hidrlise de ATP e o bombeamento de protes para fora da matriz, atravs da membrana. No entanto, quando a fora motriz alta, a reaco procede da esquerda para a direita, permitindo o fluxo de protes no sentido do gradiente de concentrao (da maior concentrao para a menor) e produzindo ATP a partir de ADP.[74] A ATP sintase um complexo proteico de grandes dimenses, em forma de cogumelo. A enzima em mamferos contm 16 subunidades e uma massa de aproximadamente 600 quilodalton.[78] A parte da enzima embebida na membrana designada FO e contm um anel de subunidades "c" e o canal de protes. O eixo e a "cabea" em forma de bola designada F1, sendo o local onde ocorre a sntese de ATP. O complexo em forma de bola na extremidade de F1 contm seis protenas de dois tipos distintos (trs subunidades e trs subunidades ); o eixo consiste numa protena (subunidade ), cuja extremidade penetra na zona das subunidades e .[79] Tanto a subunidade como a conseguem ligar nucletidos, mas apenas a subunidade catalisa a reaco de sntese do ATP. Uma outra subunidade actua como um brao lateral, estendendo-se ao longo de F1, penetrando a membrana e ligando as
Fosforilao oxidativa subunidades e base da enzima. medida que os protes atravessam a membrana atravs do canal na base da ATP sintase, FO entra em movimento de rotao.[80] Esta rotao poder ser causada por mudanas no estado de ionizao de aminocidos no anel de subunidades "c", o que poder causar interaces electrostticas que propulsionam o anel.[81] Este anel em rotao, por sua vez, fora a rotao do eixo central (subunidade ) dentro das subunidades e ; estas no entram em rotao por se encontrarem fixas pelo brao lateral, que actua como um estator. o movimento da subunidade que providencia a energia necessria para os centros activos das subunidades sofrerem alteraes que permitam a produo e libertao de ATP.[14] Esta reaco de sntese de ATP designada em Ingls como binding change mechanism (algo como "mecanismo de ligao-modificao") e consiste na modificao cclica do centro activo de cada subunidade em trs estados.[11] No estado "aberto", o ADP e o fosfato entram no centro activo. A protena muda de conformao capturando as molculas e liga-as de forma fraca (estado de ligao fraca). A enzima muda ento novamente de conformao e fora o encontro entre estas molculas (estado "fechado"), em que o centro activo liga a recm-produzida molcula de ATP com alta afinidade. O centro activo volta ento ao estado "aberto", permitindo a libertao da molcula de ATP e podendo voltar a ligar ADP e fosfato. Nalgumas bactrias e arqueas, o movimento de ies sdio, no de protes, atravs da membrana que potencia a sntese de ATP.[82] [83] Arqueas como as pertencentes ao gnero Methanococcus contm tambm a A1Ao sintase, uma forma da enzima que contm protenas com muito pouca semelhana a nvel da estrutura primria (sequncia de aminocidos) com subunidades de outras ATP sintases bacterianas e eucariticas. possvel que, nalgumas espcies, esta forma da enzima seja uma ATP sintase especializada no transporte de sdio,[84] embora tal no seja obrigatoriamente verdadeiro em todos os casos.[83]
10
Espcies reactivas de oxignioO dioxignio (oxignio molecular) um aceitador terminal de electres ideal, por ser um agente oxidante forte. A reduo do dioxignio pode originar intermedirios potencialmente danosos.[85] Embora a transferncia de quatro protes e quatro electres reduza o dioxignio a gua, uma espcie qumica incua, a transferncia de um ou dois electres produz o anio radical superxido e o perxido de hidrognio.
Estas espcies reactivas de oxignio e os seus produtos de reaco, tais como o radical hidroxilo, so muito danosos para as clulas, pois oxidam protenas e lpidos membranares e causam mutaes no DNA. Estes danos celulares podem contribuir para determinadas patologias e pensa-se que estejam envolvidos no processo de envelhecimento.[86] [87] O complexo da citocromo c oxidase muito eficiente na reduo de dioxignio a gua e produz muito poucos intermedirios parcialmente reduzidos. No entanto, so produzidas pequenas quantidades de superxido e perxido na cadeia de transporte de electres.[88] de particular importncia a reduo da coenzima Q10 no complexo III, quando existe a formao da ubisemiquinona, um radical livre muito reactivo e instvel que pode por vezes "escoar" alguns electres directamente para o oxignio, produzindo superxido.[89] Para diminuir os efeitos das espcies reactivas de oxignio, as clulas possuem diversos sistemas antioxidantes, como a presena das vitaminas C e E e enzimas como a superxido dismutase, a catalase e peroxidases,[85] que capturam e desintoxicam as espcies reactivas e limitam os danos por elas causados.
Fosforilao oxidativa
11
InibidoresExistem diversos compostos qumicos que inibem a fosforilao oxidativa. Embora normalmente qualquer um desses compostos iniba apenas uma enzima da cadeia de transporte electrnico, a inibio de apenas um dos passos suficiente para parar toda a cadeia. Por exemplo, a presena de oligomicina inibe a ATP sintase, impedindo a passagem de protes para dentro da mitocndria.[90] Tal resulta na inoperncia das bombas de protes, j que o gradiente de concentrao protnica se torna demasiado forte para ser superado. O NADH deixa ento de ser oxidado, o que pra o funcionamento do ciclo dos cidos tricarboxlicos, pois a concentrao de NAD+ cai para nveis inferiores aos necessrios para o funcionamento das enzimas desse ciclo.Compostos Cianeto Monxido de carbono Oligomicina CCCP 2,4-Dinitrofenol Rotenona Uso Venenos Efeito na fosforilao oxidativa Inibe a cadeia de transporte electrnico ao ligar o oxignio com maior afinidade que o centro FeCu do [91] citocromo c oxidase, evitando a reduo do dioxignio.
Antibitico Inibe a ATP sintase ao bloquear o fluxo de protes atravs da subunidade F .[90] O Venenos Ionforos que perturbam o gradiente de protes ao transportar protes atravs da membrana mitocondrial interna, [92] desacoplando ento o bombeamento de protes da sntese de ATP. Evita a transferncia de electres do complexo I para a ubiquinona ao bloquear o local de ligao da [93] ubiquinona.
Pesticida
Nem todos os inibidores da fosforilao oxidativa so toxinas. No tecido adiposo castanho existem canais protnicos regulados designados protenas de desacoplamento que conseguem fazer o desacoplamento da respirao e sntese de ATP.[94] Este um tipo de respirao rpida que produz calor e de particular importncia como forma de manter a temperatura corporal em animais em hibernao, embora tais protenas possam tambm ter uma funo mais geral nas respostas ao stress celular.[95]
Referncias[1] [2] [3] [4] Harden A, Young WJ. (1906). "The alcoholic ferment of yeast-juice". Proc. R. Soc. (Lond.) B (77): 40520. Kalckar HM (1974). "Origins of the concept oxidative phosphorylation". Mol. Cell. Biochem. 5 (12): 5563. PMID 4279328. Lipmann F, (1941). "Metabolic generation and utilization of phosphate bond energy". Adv Enzymol 1: 99162. Friedkin M, Lehninger AL. (1949). " Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen (http:/ / www. jbc. org/ cgi/ reprint/ 178/ 2/ 611)". J. Biol. Chem. 178 (2): 61123. [5] Slater EC. (1953). "Mechanism of Phosphorylation in the Respiratory Chain". Nature 172 (4387): 975. DOI: 10.1038/172975a0 (http:/ / dx. doi. org/ 10. 1038/ 172975a0). [6] Mitchell P. (1961). "Coupling of Phosphorylation to Electron and Hydrogen Transfer by a Chemi-Osmotic type of Mechanism". Nature 191 (4784): 144. PMID 13771349. [7] Milton H. Saier Jr. Peter Mitchell and the Vital Force (http:/ / www-biology. ucsd. edu/ ~msaier/ transport/ petermitchell/ MitchellFrame-1. html). Pgina visitada em 2007-08-23. [8] Mitchell, Peter (1978). David Keilin's Respiratory Chain Concept and Its Chemiosmotic Consequences (http:/ / nobelprize. org/ nobel_prizes/ chemistry/ laureates/ 1978/ mitchell-lecture. pdf) (Pdf). Nobel lecture. Nobel Foundation. Pgina visitada em 2007-07-21. [9] Pullman ME, Penefsky HS, Datta A, and Racker E. (1960). " Partial Resolution of the Enzymes Catalyzing Oxidative Phosphorylation. I. Purification and Properties of Soluble, Dinitrophenol-stimulated Adenosine Triphosphatase (http:/ / www. jbc. org/ cgi/ reprint/ 235/ 11/ 3322)". J. Biol. Chem. 235 (11): 33223329. [10] Boyer PD, Cross RL, Momsen W (1973). " A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=4517936)". Proc. Natl. Acad. Sci. U.S.A. 70 (10): 28379. PMID 4517936. [11] Gresser MJ, Myers JA, Boyer PD (1982). " Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model (http:/ / www. jbc. org/ cgi/ reprint/ 257/ 20/ 12030)". J. Biol. Chem. 257 (20): 120308. PMID 6214554. [12] The Nobel Prize in Chemistry 1997 (http:/ / nobelprize. org/ nobel_prizes/ chemistry/ laureates/ 1997/ ). Nobel Foundation. Pgina visitada em 2007-07-21. [13] Mitchell P, Moyle J (1967). "Chemiosmotic hypothesis of oxidative phosphorylation". Nature 213 (5072): 1379. PMID 4291593.
Fosforilao oxidativa[14] Dimroth P, Kaim G, Matthey U (2000). " Crucial role of the membrane potential for ATP synthesis by F(1)F(o) ATP synthases (http:/ / jeb. biologists. org/ cgi/ reprint/ 203/ 1/ 51)". J. Exp. Biol. 203 (Pt 1): 519. PMID 10600673. [15] Schultz B, Chan S (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes". Annu Rev Biophys Biomol Struct 30: 2365. PMID 11340051. [16] Rich PR (2003). " The molecular machinery of Keilin's respiratory chain (http:/ / www. biochemsoctrans. org/ bst/ 031/ 1095/ bst0311095. htm)". Biochem. Soc. Trans. 31 (Pt 6): 1095105. PMID 14641005. [17] Porter RK, Brand MD (1995). " Mitochondrial proton conductance and H+/O ratio are independent of electron transport rate in isolated hepatocytes (http:/ / www. pubmedcentral. nih. gov/ picrender. fcgi?artid=1135905& blobtype=pdf)". Biochem. J. 310 ( Pt 2): 37982. PMID 7654171. [18] Mathews FS (1985). "The structure, function and evolution of cytochromes". Prog. Biophys. Mol. Biol. 45 (1): 156. PMID 3881803. [19] Wood PM (1983). "Why do c-type cytochromes exist?". FEBS Lett. 164 (2): 2236. PMID 6317447. [20] Crane FL (2001). " Biochemical functions of coenzyme Q10 (http:/ / www. jacn. org/ cgi/ content/ full/ 20/ 6/ 591)". Journal of the American College of Nutrition 20 (6): 5918. PMID 11771674. [21] Mitchell P (1979). "Keilin's respiratory chain concept and its chemiosmotic consequences". Science 206 (4423): 114859. PMID 388618. [22] Sballe B, Poole RK (1999). " Microbial ubiquinones: multiple roles in respiration, gene regulation and oxidative stress management (http:/ / mic. sgmjournals. org/ cgi/ reprint/ 145/ 8/ 1817. pdf)". Microbiology (Reading, Engl.) 145 ( Pt 8): 181730. PMID 10463148. [23] Johnson D, Dean D, Smith A, Johnson M (2005). "Structure, function, and formation of biological iron-sulfur clusters". Annu Rev Biochem 74: 24781. PMID 15952888. [24] Page CC, Moser CC, Chen X, Dutton PL (1999). "Natural engineering principles of electron tunnelling in biological oxidation-reduction". Nature 402 (6757): 4752. PMID 10573417. [25] Leys D, Scrutton NS (2004). "Electrical circuitry in biology: emerging principles from protein structure". Curr. Opin. Struct. Biol. 14 (6): 6427. PMID 15582386. [26] Boxma B, de Graaf RM, van der Staay GW, et al (2005). "An anaerobic mitochondrion that produces hydrogen". Nature 434 (7029): 749. PMID 15744302. [27] Hirst J (2005). " Energy transduction by respiratory complex I--an evaluation of current knowledge (http:/ / www. biochemsoctrans. org/ bst/ 033/ 0525/ 0330525. pdf)". Biochem. Soc. Trans. 33 (Pt 3): 5259. PMID 15916556. [28] Lenaz G, Fato R, Genova M, Bergamini C, Bianchi C, Biondi A (2006). "Mitochondrial Complex I: structural and functional aspects". Biochim Biophys Acta 1757 (910): 140620. PMID 16828051. [29] Sazanov L.A., Hinchliffe P. (2006) Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus. Science 311, 1430-1436 [30] Baranova EA, Holt PJ, Sazanov LA (2007). "Projection structure of the membrane domain of Escherichia coli respiratory complex I at 8 A resolution". J. Mol. Biol. 366 (1): 14054. PMID 17157874. [31] Friedrich T, Bttcher B (2004). "The gross structure of the respiratory complex I: a Lego System". Biochim. Biophys. Acta 1608 (1): 19. PMID 14741580. [32] Brandt U, Kerscher S, Drse S, Zwicker K, Zickermann V (2003). "Proton pumping by NADH:ubiquinone oxidoreductase. A redox driven conformational change mechanism?". FEBS Lett. 545 (1): 917. PMID 12788486. [33] Cecchini G (2003). "Function and structure of complex II of the respiratory chain". Annu Rev Biochem 72: 77109. PMID 14527321. [34] Yankovskaya V., Horsefield R., Tornroth S., Luna-Chavez C., Miyoshi H., Leger C., Byrne B., Cecchini G., Iwata S. (2003) Architecture of succinate dehydrogenase and reactive oxygen species generation. Science 299, 700-704 [35] Horsefield R, Iwata S, Byrne B (2004). "Complex II from a structural perspective". Curr. Protein Pept. Sci. 5 (2): 10718. PMID 15078221. [36] Kita K, Hirawake H, Miyadera H, Amino H, Takeo S (2002). "Role of complex II in anaerobic respiration of the parasite mitochondria from Ascaris suum and Plasmodium falciparum". Biochim. Biophys. Acta 1553 (12): 12339. PMID 11803022. [37] Painter HJ, Morrisey JM, Mather MW, Vaidya AB (2007). "Specific role of mitochondrial electron transport in blood-stage Plasmodium falciparum". Nature 446 (7131): 8891. PMID 17330044. [38] Ramsay RR, Steenkamp DJ, Husain M (1987). "Reactions of electron-transfer flavoprotein and electron-transfer flavoprotein: ubiquinone oxidoreductase". Biochem. J. 241 (3): 88392. PMID 3593226. [39] Zhang J, Frerman FE, Kim JJ (2006). "Structure of electron transfer flavoprotein-ubiquinone oxidoreductase and electron transfer to the mitochondrial ubiquinone pool". Proc. Natl. Acad. Sci. U.S.A. 103 (44): 162127. DOI: 10.1073/pnas.0604567103 (http:/ / dx. doi. org/ 10. 1073/ pnas. 0604567103). PMID 17050691. [40] Ikeda Y, Dabrowski C, Tanaka K (1983). " Separation and properties of five distinct acyl-CoA dehydrogenases from rat liver mitochondria. Identification of a new 2-methyl branched chain acyl-CoA dehydrogenase (http:/ / www. jbc. org/ cgi/ reprint/ 258/ 2/ 1066)". J. Biol. Chem. 258 (2): 106676. PMID 6401712. [41] Ruzicka FJ, Beinert H (1977). " A new iron-sulfur flavoprotein of the respiratory chain. A component of the fatty acid beta oxidation pathway (http:/ / www. jbc. org/ cgi/ reprint/ 252/ 23/ 8440. pdf)". J. Biol. Chem. 252 (23): 84405. PMID 925004. [42] Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ (2005). " The critical role of Arabidopsis electron-transfer flavoprotein:ubiquinone oxidoreductase during dark-induced starvation (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgitool=pubmed& pubmedid=16055629)". Plant Cell 17 (9): 2587600. PMID 16055629. [43] Berry E, Guergova-Kuras M, Huang L, Crofts A (2000). "Structure and function of cytochrome bc complexes". Annu Rev Biochem 69: 100575. PMID 10966481.
12
Fosforilao oxidativa[44] Crofts AR (2004). "The cytochrome bc1 complex: function in the context of structure". Annu. Rev. Physiol. 66: 689733. PMID 14977419. [45] Iwata S, Lee JW, Okada K, et al (1998). "Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex". Science 281 (5373): 6471. PMID 9651245. [46] Trumpower BL (1990). " The protonmotive Q cycle. Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex (http:/ / www. jbc. org/ cgi/ reprint/ 265/ 20/ 11409. pdf)". J. Biol. Chem. 265 (20): 1140912. PMID 2164001. [47] Hunte C, Palsdottir H, Trumpower BL (2003). "Protonmotive pathways and mechanisms in the cytochrome bc1 complex". FEBS Lett. 545 (1): 3946. PMID 12788490. [48] Calhoun M, Thomas J, Gennis R (1994). "The cytochrome oxidase superfamily of redox-driven proton pumps". Trends Biochem Sci 19 (8): 32530. PMID 7940677. [49] Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S. (1996). "TThe whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A.". Science 272 (5265): 113644. PMID 8638158. [50] Yoshikawa S, Muramoto K, Shinzawa-Itoh K, et al (2006). "Proton pumping mechanism of bovine heart cytochrome c oxidase". Biochim. Biophys. Acta 1757 (910): 11106. PMID 16904626. [51] Rasmusson AG, Soole KL, Elthon TE (2004). "Alternative NAD(P)H dehydrogenases of plant mitochondria". Annual review of plant biology 55: 2339. PMID 15725055. [52] Menz RI, Day DA (1996). " Purification and characterization of a 43-kDa rotenone-insensitive NADH dehydrogenase from plant mitochondria (http:/ / www. jbc. org/ cgi/ content/ full/ 271/ 38/ 23117)". J. Biol. Chem. 271 (38): 2311720. PMID 8798503. [53] McDonald A, Vanlerberghe G (2004). "Branched mitochondrial electron transport in the Animalia: presence of alternative oxidase in several animal phyla". IUBMB Life 56 (6): 33341. PMID 15370881. [54] Sluse FE, Jarmuszkiewicz W (1998). "Alternative oxidase in the branched mitochondrial respiratory network: an overview on structure, function, regulation, and role". Braz. J. Med. Biol. Res. 31 (6): 73347. PMID 9698817. [55] Moore AL, Siedow JN (1991). "The regulation and nature of the cyanide-resistant alternative oxidase of plant mitochondria". Biochim. Biophys. Acta 1059 (2): 12140. PMID 1883834. [56] Vanlerberghe GC, McIntosh L (1997). "Alternative oxidase: From Gene to Function" 48: 70334. PMID 15012279. [57] Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A (1997). "Transcript levels of tandem-arranged alternative oxidase genes in rice are increased by low temperature". Gene 203 (2): 1219. PMID 9426242. [58] Maxwell DP, Wang Y, McIntosh L (1999). " The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells (http:/ / www. pnas. org/ cgi/ content/ full/ 96/ 14/ 8271)". Proc. Natl. Acad. Sci. U.S.A. 96 (14): 82716. PMID 10393984. [59] Heinemeyer J, Braun HP, Boekema EJ, Kouril R (2007). "A structural model of the cytochrome C reductase/oxidase supercomplex from yeast mitochondria". J. Biol. Chem. 282 (16): 122408. PMID 17322303. [60] Schgger H, Pfeiffer K (2000). " Supercomplexes in the respiratory chains of yeast and mammalian mitochondria (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=10775262)". EMBO J. 19 (8): 177783. PMID 10775262. [61] Schgger H (2002). "Respiratory chain supercomplexes of mitochondria and bacteria". Biochim. Biophys. Acta 1555 (13): 1549. PMID 12206908. [62] Schgger H, Pfeiffer K (2001). " The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes (http:/ / www. jbc. org/ cgi/ content/ full/ 276/ 41/ 37861)". J. Biol. Chem. 276 (41): 378617. PMID 11483615. [63] Gupte S, Wu ES, Hoechli L, et al (1984). " Relationship between lateral diffusion, collision frequency, and electron transfer of mitochondrial inner membrane oxidation-reduction components (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=6326133)". Proc. Natl. Acad. Sci. U.S.A. 81 (9): 260610. PMID 6326133. [64] Nealson KH (1999). "Post-Viking microbiology: new approaches, new data, new insights". Origins of life and evolution of the biosphere : the journal of the International Society for the Study of the Origin of Life 29 (1): 7393. PMID 11536899. [65] Ingledew WJ, Poole RK (1984). "The respiratory chains of Escherichia coli". Microbiol. Rev. 48 (3): 22271. PMID 6387427. [66] Schfer G, Engelhard M, Mller V (1999). " Bioenergetics of the Archaea (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=10477309)". Microbiol. Mol. Biol. Rev. 63 (3): 570-620. PMID 10477309. [67] Unden G, Bongaerts J (1997). "Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors". Biochim. Biophys. Acta 1320 (3): 21734. PMID 9230919. [68] Cecchini G, Schrder I, Gunsalus RP, Maklashina E (2002). "Succinate dehydrogenase and fumarate reductase from Escherichia coli". Biochim. Biophys. Acta 1553 (12): 14057. PMID 11803023. [69] Freitag A, Bock E (1990). "Energy conservation in Nitrobacter". FEMS Microbiology Letters 66 (13): 157&ndash:62. DOI: 10.1111/j.1574-6968.1990.tb03989.x (http:/ / dx. doi. org/ 10. 1111/ j. 1574-6968. 1990. tb03989. x). [70] Starkenburg SR, Chain PS, Sayavedra-Soto LA, et al (2006). " Genome sequence of the chemolithoautotrophic nitrite-oxidizing bacterium Nitrobacter winogradskyi Nb-255 (http:/ / aem. asm. org/ cgi/ content/ full/ 72/ 3/ 2050?view=long& pmid=16517654)". Appl. Environ. Microbiol. 72 (3): 205063. PMID 16517654. [71] Yamanaka T, Fukumori Y (1988). "The nitrite oxidizing system of Nitrobacter winogradskyi". FEMS Microbiol. Rev. 4 (4): 25970. PMID 2856189. [72] Iuchi S, Lin EC (1993). "Adaptation of Escherichia coli to redox environments by gene expression". Mol. Microbiol. 9 (1): 915. PMID 8412675.
13
Fosforilao oxidativa[73] Calhoun MW, Oden KL, Gennis RB, de Mattos MJ, Neijssel OM (1993). " Energetic efficiency of Escherichia coli: effects of mutations in components of the aerobic respiratory chain (http:/ / jb. asm. org/ cgi/ reprint/ 175/ 10/ 3020. pdf)". J. Bacteriol. 175 (10): 30205. PMID 8491720. [74] Boyer PD (1997). "The ATP synthase--a splendid molecular machine". Annu. Rev. Biochem. 66: 71749. PMID 9242922. [75] Van Walraven HS, Strotmann H, Schwarz O, Rumberg B (1996). "The H+/ATP coupling ratio of the ATP synthase from thiol-modulated chloroplasts and two cyanobacterial strains is four". FEBS Lett. 379 (3): 309-13. PMID 8603713. [76] Yoshida M, Muneyuki E, Hisabori T (2001). "ATP synthase--a marvellous rotary engine of the cell". Nat. Rev. Mol. Cell Biol. 2 (9): 669-77. PMID 11533724. [77] Schemidt RA, Qu J, Williams JR, Brusilow WS (1998). " Effects of carbon source on expression of F0 genes and on the stoichiometry of the c subunit in the F1F0 ATPase of Escherichia coli (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=9620972)". J. Bacteriol. 180 (12): 3205-8. PMID 9620972. [78] Rubinstein JL, Walker JE, Henderson R (2003). " Structure of the mitochondrial ATP synthase by electron cryomicroscopy (http:/ / www. pubmedcentral. nih. gov/ picrender. fcgi?artid=291849& blobtype=pdf)". EMBO J. 22 (23): 618292. PMID 14633978. [79] Leslie AG, Walker JE (2000). " Structural model of F1-ATPase and the implications for rotary catalysis (http:/ / www. pubmedcentral. nih. gov/ picrender. fcgi?artid=1692760& blobtype=pdf)". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 355 (1396): 46571. PMID 10836500. [80] Noji H, Yoshida M (2001). " The rotary machine in the cell, ATP synthase (http:/ / www. jbc. org/ cgi/ content/ full/ 276/ 3/ 1665)". J. Biol. Chem. 276 (3): 1665-8. PMID 11080505. [81] Capaldi R, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem Sci 27 (3): 15460. PMID 11893513. [82] Dimroth P (1994). "Bacterial sodium ion-coupled energetics". Antonie Van Leeuwenhoek 65 (4): 38195. PMID 7832594. [83] Becher B, Mller V (1994). " Delta mu Na+ drives the synthesis of ATP via a delta mu Na(+)-translocating F1F0-ATP synthase in membrane vesicles of the archaeon Methanosarcina mazei G1 (http:/ / www. pubmedcentral. nih. gov/ articlerender. fcgi?tool=pubmed& pubmedid=8169202)". J. Bacteriol. 176 (9): 254350. PMID 8169202. [84] Mller V (2004). "An exceptional variability in the motor of archael A1A0 ATPases: from multimeric to monomeric rotors comprising 613 ion binding sites". J. Bioenerg. Biomembr. 36 (1): 11525. PMID 15168615. [85] Davies K (1995). "Oxidative stress: the paradox of aerobic life". Biochem Soc Symp 61: 131. PMID 8660387. [86] Rattan SI (2006). "Theories of biological aging: genes, proteins, and free radicals". Free Radic. Res. 40 (12): 12308. PMID 17090411. [87] Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J (2007). "Free radicals and antioxidants in normal physiological functions and human disease". Int. J. Biochem. Cell Biol. 39 (1): 4484. PMID 16978905. [88] Raha S, Robinson B (2000). "Mitochondria, oxygen free radicals, disease and ageing". Trends Biochem Sci 25 (10): 5028. PMID 11050436. [89] Finkel T, Holbrook NJ (2000). "Oxidants, oxidative stress and the biology of ageing". Nature 408 (6809): 23947. PMID 11089981. [90] Joshi S, Huang YG (1991). "ATP synthase complex from bovine heart mitochondria: the oligomycin sensitivity conferring protein is essential for dicyclohexyl carbodiimide-sensitive ATPase". Biochim. Biophys. Acta 1067 (2): 2558. PMID 1831660. [91] Tsubaki M (1993). "Fourier-transform infrared study of cyanide binding to the Fea3-CuB binuclear site of bovine heart cytochrome c oxidase: implication of the redox-linked conformational change at the binuclear site". Biochemistry 32 (1): 16473. PMID 8380331. [92] Heytler PG (1979). "Uncouplers of oxidative phosphorylation". Meth. Enzymol. 55: 46242. PMID 156853. [93] Lambert AJ, Brand MD (2004). " Inhibitors of the quinone-binding site allow rapid superoxide production from mitochondrial NADH:ubiquinone oxidoreductase (complex I) (http:/ / www. jbc. org/ cgi/ content/ full/ 279/ 38/ 39414)". J. Biol. Chem. 279 (38): 3941420. PMID 15262965. [94] Ricquier D, Bouillaud F (2000). " The uncoupling protein homologues: UCP1, UCP2, UCP3, StUCP and AtUCP (http:/ / www. pubmedcentral. nih. gov/ picrender. fcgi?artid=1220743& blobtype=pdf)". Biochem. J. 345 Pt 2: 16179. PMID 10620491. [95] Boreck J, Vercesi AE (2005). "Plant uncoupling mitochondrial protein and alternative oxidase: energy metabolism and stress". Biosci. Rep. 25 (3-4): 27186. PMID 16283557.
14
Bibliografia recomendadaIntrodutria Nelson DL. Lehninger Principles of Biochemistry. 4th eded. [S.l.]:W. H. Freeman, 2004. ISBN 0-716-74339-6 Schneider ED. Into the Cool: Energy Flow, Thermodynamics and Life. 1st eded. [S.l.]:University of Chicago Press, 2006. ISBN 0-226-73937-6 Avanada Nicholls DG. Bioenergetics 3. 1st eded. [S.l.]:Academic Press, 2002. ISBN 0-125-18121-3 Haynie D. Biological Thermodynamics. 1st eded. [S.l.]:Cambridge University Press, 2001. ISBN 0-521-79549-4 Rajan SS. Introduction to Bioenergetics. 1st eded. [S.l.]:Anmol, 2003. ISBN 8-126-11364-2
Fosforilao oxidativa Wikstrom M (Ed). Biophysical and Structural Aspects of Bioenergetics. 1st eded. [S.l.]:Royal Society of Chemistry, 2005. ISBN 0-854-04346-2
15
Ligaes externasGerais Diagramas animados ilustrando a fosforilao oxidativa (http://www.wiley.com/legacy/college/boyer/ 0470003790/animations/electron_transport/electron_transport.htm) (em ingls): Wiley and Co, Concepts in Biochemistry Fosforilao oxidativa (http://www.gwu.edu/~mpb/oxidativephos.htm) (em ingls): Metabolic Pathways of Biochemistry, a referncia online sobre metabolismo para estudantes e professores na Universidade George Washington ATP sintase o motor rotatrio da clula (http://www.res.titech.ac.jp/~seibutu/) (em ingls): breve introduo, incluindo vdeos de microfotografias da enzima em rotao, no Tokyo Institute of Technology Lies online de biofsica (http://www.life.uiuc.edu/crofts/bioph354/) (em ingls): Antony Crofts, Universidade de Illinois em Urbana-Champaign Sobre as estruturas Animaes da ATP sintase (http://nature.berkeley.edu/~hongwang/Project/ATP_synthase/) (em ingls) Hongyun Wang e George Oster, Universidade da Califrnia em Berkeley Molcula do ms PDB: ATP sintase (http://www.rcsb.org/pdb/static.do?p=education_discussion/molecule_of_the_month/ pdb72_1.html) (em ingls) Citocromo c (http://www.rcsb.org/pdb/static.do?p=education_discussion/molecule_of_the_month/ pdb36_1.html) (em ingls) Citocromo c oxidase (http://www.rcsb.org/pdb/static.do?p=education_discussion/ molecule_of_the_month/pdb5_1.html) (em ingls) Modelos moleculares interactivos na Universidade Fernando Pessoa: NADH desidrogenase (http://www2.ufp.pt/~pedros/anim/2frame-i.htm) (em portugus) Succinato desidrogenase (http://www2.ufp.pt/~pedros/anim/2frame-ii.htm) (em portugus) Coenzima Q - citocromo c redutase (http://www2.ufp.pt/~pedros/anim/2frame-iii.htm) (em portugus) Citocromo c oxidase (http://www2.ufp.pt/~pedros/anim/2frame-iv.htm) (em portugus)
Fontes e Editores da Pgina
16
Fontes e Editores da PginaFosforilao oxidativa Fonte: http://pt.wikipedia.org/w/index.php?oldid=27321497 Contribuidores: Alchimista, Ana couto, Beria, Bons, Brunoff, Comte0, GOE, Giro720, Joo Sousa, Lijealso, Luabio, MachoCarioca, Marcos Elias de Oliveira Jnior, Mateus RM, PatrciaR, Pomonews, Robertogilnei, Santana-freitas, Tiago Becerra Paolini, Viniciusmc, Vitorbenigno, 21 edies annimas
Fontes, Licenas e Editores da ImagemFicheiro:Mitochondrial electron transport chainEtc4 pt.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Mitochondrial_electron_transport_chainEtc4_pt.svg Licena: Public Domain Contribuidores: Myself, based on Fvasconcellos' vectorization of TimVickers' en:Image:Etc4.png. Ficheiro:Ubiquinoneubiquinol conversion.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Ubiquinoneubiquinol_conversion.svg Licena: Public Domain Contribuidores: Fvasconcellos 03:07, 12 September 2007 (UTC) Ficheiro:Complex I pt.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Complex_I_pt.svg Licena: Public Domain Contribuidores: Myself, based on Fvasconcellos vectorization. Ficheiro:Complex II pt.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Complex_II_pt.svg Licena: Public Domain Contribuidores: Myself, based on Fvasconcellos vectorization. Ficheiro:Complex III reaction pt.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Complex_III_reaction_pt.svg Licena: Public Domain Contribuidores: Myself, based on Fvasconcellos vectorization. Ficheiro:Complex IV pt.svg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Complex_IV_pt.svg Licena: Public Domain Contribuidores: Myself, based on Fvasconcellos vectorization. Ficheiro:Atpsynthase.jpg Fonte: http://pt.wikipedia.org/w/index.php?title=Ficheiro:Atpsynthase.jpg Licena: Creative Commons Attribution-Sharealike 2.5 Contribuidores: -
LicenaCreative Commons Attribution-Share Alike 3.0 Unported //creativecommons.org/licenses/by-sa/3.0/