UBA VII – Genética Molecular Genética Molecular e Humana Teórica 5.
Identificando a programação genética da célula através do uso dos microchips de DNA Ana...
-
Upload
natalia-rosa-estrela -
Category
Documents
-
view
214 -
download
1
Transcript of Identificando a programação genética da célula através do uso dos microchips de DNA Ana...

Identificando a programação genética da célula através do
uso dos microchips de DNA
Ana Carolina DeckmannLaboratório de Genômica e Expressão
Depto. Genética e Evolução - Instituto de Biologia - Unicamp
Minicurso “Da Genômica à Biotecnologia” - Minicurso “Da Genômica à Biotecnologia” - IV Semana de Química/ 2004IV Semana de Química/ 2004

Projetos Genoma: o que nos dizem?
• Projetos Genoma: deciframento das sequências de DNA, sua organização e estrutura.
• Ao decifrar um genoma, resta a dúvida: se todas as células de um organismo carregam o mesmo genoma, O QUE as torna diferentes umas das outras???

Estudos funcionais

Introdução aos chips de Introdução aos chips de DNADNA

• Os chips de DNA fornecem um método conveniente para explorar o genoma em um nível molecular.
• Análise de milhares de informações genéticas em um único experimento.
• A utilização desta técnica suporta muitas possibilidades e novas aplicações surgem diariamente.
Os Biochips e sua importância

Atualmente, as principais aplicações são:
• Genômica funcional (um genoma)
• Genômica comparativa (vários genomas)
• Genotipagem (SNPs)
• Estudo da cromatina (ChIP-ON-CHIP)
Os Biochips e sua importância

Fundamentos teóricosFundamentos teóricos

•PROPRIEDADE 1) a estrutura em dupla hélice é mantida unida pela complementariedade entre as bases nucleotídicas.
Fundamentos Teóricos

Fundamentos Teóricos
• Consequência: hibridização
DNA fita dupla
DNA desnaturado
hibridização

• PROPRIEDADE 2) a produção de proteínas depende de etapas intermediárias em que o DNA é transcrito em moléculas de RNAm COMPLEMENTARES à sequência do gene!
Fundamentos Teóricos
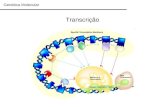
DOGMA CENTRAL DA BIOLOGIA MOLECULAR

• Consequência: a função gênica pode ser estudada através das etapas de transcrição!
• COMO?
• Medindo quantos híbridos se formam entre DNA e mRNA, e assim quantificando a expressão gênica numa situação de interesse, p.ex., câncer!
• COMO QUANTIFICAR A FORMAÇÃO DE HÍBRIDOS?
Fundamentos Teóricos

transcrição reversa na presença de bases marcadas
CGATTAGC
cDNA marcado
genoma
gene A
transcrição
expressão do gene A
DNACGATTAGC
RNAmGCUAAUCG
transcrição reversa
cDNACGATTAGC
tradução
Proteína A
Fundamentos Teóricos
célula

• DNA marcado por radioatividade
Estudos clássicos de expressão gênica (caso a caso). Ex: Northern blotting.
Fundamentos Teóricos
1) isolar as mensagens produzidas em uma célula (mRNA) e imobilizá-las em um suporte sólido.
2) marcar a sequência do gene de interesse durante a reação de polimerização.
3) Hibridizar!

C 1h 3h 6h 12h 48h
Gene -MHC
rRNA 28S
rRNA 18S
Northern blotting
Fundamentos Teóricos

1) isolar um grande número de genes (BIBLIOTECA DE cDNA) e organizá-los lado a lado em um suporte sólido.
2) marcar as mensagens produzidas em uma célula com radioatividade ou fluorescência durante a reação de transcrição reversa.
3) Hibridizar!
Fundamentos Teóricos
• DNA marcado por fluorescência: macro- e microarrays

Chip Cp Chip Rn
Fundamentos Teóricos

Fabricação dos biochips Fabricação dos biochips de DNAde DNA

BIBLIOTECA DE cDNA
Placas 384 fita molde
fita molde
produtos
X ciclos
AMPLIFICAÇÃO
Eletroforese dos insertos da biblioteca de cDNA de coração de rato.
PRODUÇÃO DOS MICROARRANJOS

Fonte: Genomics Solutions. http://www.genomicsolutions.com
Flexys Robot (Perkin Elmer)
Deposição automatizada dos clones em posições pré-determinadas.
ESPOTAGEM
Organização da biblioteca em microplacas 384.
PRODUÇÃO DOS MICROARRANJOS

SÍNTESE DAS SONDAS FLUORESCENTES
MARCAÇÃO DAS SONDAS
C 1 3 6 12 48
RNA total
RNA[controle]
Cy3
Cy5
x RNA[exp]
PRODUÇÃO DOS MICROARRANJOS

MICROARRANJO
HIBRIDAÇÃO
SPOT
DNA fita simples Hibridação com as sondas
desnaturadas
SCANNER
GeneTAC 2000 (Perkin Elmer)Fonte: Genomics Solutions. http://www.genomicsolutions.com
GRADE
PRODUÇÃO DOS MICROARRANJOS

EXCITAÇÃO DAS SONDAS E CAPTURA DOS SINAIS
fonte de luz
570nm
670nm
absorção emissão
550nm
Cy3
649nm
Cy5
PRODUÇÃO DOS MICROARRANJOS

Esquema do endereçamento dos pontos na imagem.
abcd
ef
gh
ij
k
A
B
C
D
E
F
G
H
1 2 3 4PROCESSAMENTO DA IMAGEM
A1.a2
Estimativa computacional das intensidades de fluorescência emitidas por Cy3 e por Cy5 e cálculo da razão.
Address
Total Intensity (Cy3)
Total Intensity (Cy5) Ratio
Plate Name
Plate Well
A1.a1 40032 44144 1.86 1 A1A1.a10 1355840 537744 0.67 2 I17A1.a11 17600 38712 3.71 1 A1A1.a2 1309408 329272 0.42 2 I17A1.a3 106576 29248 0.46 2 A21A1.a5 35560 33472 1.59 1 A1A1.a6 1269856 505080 0.67 2 I17A1.a7 117472 5912 0.08 2 A21A1.a8 64224 18032 0.47 3 I17A1.a9 107552 20904 0.33 2 A21
1 2 3 4 5 6 7 8 9 10 11
A1.a7
PRODUÇÃO DOS MICROARRANJOS

Geração de dados de Expressão Gênica

intensidade Cy5 intensidade Cy3 = razão de expressão
Razão > 1(gene induzido Cy5)
Razão = 1 Razão < 1(gene induzido Cy3)
Geração de dados de Expressão Gênica

Data miningData mining

• Ao considerar cada um dos n chips produzidos como uma das n observações para um dado gene, é possível aplicar técnicas de análise multivariada para extrair significados biológicos dos experimentos de microarranjos de DNA.
• Clustering algorithms: reconhecer padrões nas distribuições de dados, normalmente através de métricas de distância.
• Tipos:• Aglomerativos: Hierarchical• Divisivos: k-means, SOMs• Outros: Principal Component Analysis (PCA)
Data mining

Data mining
• A representação gráfica assume a forma de uma curva normal. • A transformação logarítmica permite representar repressões e induções de igual magnitude como valores numericamente iguais mas de sinais diferentes.•Eisen e col. (1998): LOG positivo = vermelho (gene induzido) LOG negativo = verde (gene reprimido)
Razão 0,5 ------ Log0,5 = -1 Razão 1,0 ------ Log1 = 0
Razão 2,0 ------ Log2 = 1
Distância=0,5
Distância=1,0
Distância=1,0
Distância=1,0

Data mining
Gen
e 1
Gen
e 2
Gen
e 3
Gen
e 4
Gen
e 5
Gen
e 6
Gene1 0 1.5 1.2 0.25 0.75 1.4
Gene2 1.5 0 1.3 0.55 2.0 1.5 Gene3 1.2 1.3 0 1.3 0.75 0.3Gene4 0.25 0.55 1.3 0 0.25 0.4 Gene5 0.75 2.0 0.75 0.25 0 1.2 Gene6 1.4 1.5 0.3 0.4 1.2 0
• Após o cálculo da distância, o ponto de partida dos algoritmos de clustering é gerar uma matriz de distância.
• Os elementos da matriz são distâncias entre pares (pairwise distance).

Aplicações
Cluster analysis and display of genome-wide expression patterns.
PNAS, 95 (1998)
Michael B. Eisen, Paul T. Spellman, Patrick O. Brown, and David Botstein

The Transcriptional Program of Sporulation
in Budding Yeast
Science, 282 (1998)
S. Chu, J. DeRisi, M. Eisen, J. Mulholland, D. Botstein, P. O. Brown, I. Herskowitz
Aplicações

Molecular Classification of
Cancer: Class Discovery and Class Prediction by Gene
Expression Monitoring.
Science, 286 (1999)
T. R. Golub, D. K. Slonim, P. Tamayo, et al.
Aplicações

Biochips de Biochips de oligonucleotídeosoligonucleotídeos

• Principais áreas em que a aplicação dos chips de oligonucleotídeos podem acelerar os estudos em genômica:
•Expressão gênica
•Genotipagem
•Mapeamento de bibliotecas genômicas
Biochips de Oligonucleotídeos

Técnica de microimpressão: Técnica de microimpressão: FOTOLITOGRAFIAFOTOLITOGRAFIA
• Esta técnica de fabricação in situ foi desenvolvida pela empresa americana Affymetrix, que utiliza para fabricar os GeneChips.
Biochips de Oligonucleotídeos

Biochips de Oligonucleotídeos

Biochips de Oligonucleotídeos

Biochips de Oligonucleotídeos
• Diferenciais:
1) alta densidade permite representação de genomas completos.
2) refinamento da técnica de microimpressão permite criar mutações (mismatchs) de sequências conhecidas.
3) quantificações mais precisas/ alta sensibilidade.

Biochips de Oligonucleotídeos

Biochips de Oligonucleotídeos
Extração DNA
cromossômico
Gene A
Amplificação por PCR na presença de fluoróforo
Cópias marcadas do
gene AHibridização com chip de
oligos

Biochips de Oligonucleotídeos
Gene A
MISMATCH SINALMISMATCH SINAL1A 1A C G G T A C T C G G T A C T aa G A G G A G G C T A G C T A 1 1 + +1G 1G C G G T A C T C G G T A C T gg G A G G A G G C T A G C T A 1 1 + +1C 1C C G G T A C T C G G T A C T cc G A G G A G G C T A G C T A 1 1 + +1T 1T C G G T A C T C G G T A C T tt G A G G A G G C T A G C T A 0 0 ++ ++
1A 1A C G G T A C T C G G T A C T T T aa A G A G G C T A G C T A 1 1 + +1G 1G C G G T A C T C G G T A C T T T gg A G A G G C T A G C T A 0 0 ++ ++1C 1C C G G T A C T C G G T A C T T T cc A G A G G C T A G C T A 1 1 + +1T 1T C G G T A C T C G G T A C T T T tt A G A G G C T A G C T A 1 1 + +
1A 1A C G G T A C T C G G T A C T T G T G aa G G G C T A G C T A 0 0 ++ ++1G 1G C G G T A C T C G G T A C T T G T G gg G G G C T A G C T A 1 1 + +1C 1C C G G T A C T C G G T A C T T G T G cc G G G C T A G C T A 1 1 + +1T 1T C G G T A C T C G G T A C T T G T G tt G G G C T A G C T A 1 1 + +
1A 1A C G G T A C T C G G T A C T T G A T G A aa G C T A G C T A 1 1 + +1G 1G C G G T A C T C G G T A C T T G A T G A gg G C T A G C T A 0 0 ++ ++1C 1C C G G T A C T C G G T A C T T G A T G A cc G C T AG C T A 1 1 + +1T 1T C G G T A C T C G G T A C T T G A T G A tt G C T A G C T A 1 1 + +
Padrão normalPadrão afetado
1 2 3 4
AGCT
Pessoa 1: normal

Biochips de Oligonucleotídeos
Gene A
MISMATCH SINALMISMATCH SINAL1A 1A C G G T A C T C G G T A C T aa G A G G A G G C T A G C T A 1 1 + +1G 1G C G G T A C T C G G T A C T gg G A G G A G G C T A G C T A 1 1 + +1C 1C C G G T A C T C G G T A C T cc G A G G A G G C T A G C T A 0 0 ++ ++1T 1T C G G T A C T C G G T A C T tt G A G G A G G C T A G C T A 1 1 + +
1A 1A C G G T A C T C G G T A C T T T aa A G A G G C T A G C T A 2 2 - -1G 1G C G G T A C T C G G T A C T T T gg A G A G G C T A G C T A 1 1 + +1C 1C C G G T A C T C G G T A C T T T cc A G A G G C T A G C T A 2 2 - -1T 1T C G G T A C T C G G T A C T T T tt A G A G G C T A G C T A 2 2 - -
1A 1A C G G T A C T C G G T A C T T G T G aa G G G C T A G C T A 1 1 + +1G 1G C G G T A C T C G G T A C T T G T G gg G G G C T A G C T A 2 2 - -1C 1C C G G T A C T C G G T A C T T G T G cc G G G C T A G C T A 2 2 - -1T 1T C G G T A C T C G G T A C T T G T G tt G G G C T A G C T A 2 2 - -
1A 1A C G G T A C T C G G T A C T T G A T G A aa G C T A G C T A 2 2 - -1G 1G C G G T A C T C G G T A C T T G A T G A gg G C T A G C T A 1 1 + +1C 1C C G G T A C T C G G T A C T T G A T G A cc G C T AG C T A 2 2 - -1T 1T C G G T A C T C G G T A C T T G A T G A tt G C T A G C T A 2 2 - -
Padrão normalPadrão afetado
AGCT
1 2 3 4
Pessoa 2: afetada

ChIP-on-CHIPChIP-on-CHIP
Chromatin Chromatin Imunnoprecipitation Imunnoprecipitation
microarraysmicroarrays

Chromatin Imunnoprecipitation microarrays
• Normalmente, nos biochips estão representadas as partes codificantes do genoma (ESTs, cDNAs).

Chromatin Imunnoprecipitation microarrays
• Objetivo: identificar no DNA genômico os sítios de ligação a TFs, histonas e polimerases.
• Biochips de oligonucleotídeos longos (60 pb) de ORFs e regiões intergênicas.• Representação de genomas completos (~40Mb)• Sondas: DNA imunoprecipitado x controle (precipitação sem anticorpo)
Maior sinalconfirmação
da especificidade
da ligação!
mais fragmentos do DNA
correpondente ligados nas proteína de interesse

Chromatin Imunnoprecipitation microarrays
(~1kb)(~1kb)

Projetos em andamento

LGE
Estudo do perfil da expressão gênicaglobal em leucemias linfóides agudas de
linhagens de células B e T
Aluna : Diana Azevedo QueirozOrientador: Prof. Dr. Gonçalo A. G. Pereira
Laboratório de Genômica e Expressão – Departamento de Genética e Evolução – Instituto de Biologia - Unicamp
Dissertação de Mestrado em Biologia Funcional e Molecular
Análise do Perfi l de ExpressãoGênica de Mediadores
I nflamatórios em CélulasMononucleares de Pacientes
com Trombose Venosa Prof undaAlunaAluna: Michelle: Michelle Servais Rubin Servais Rubin
OrientadoraOrientadora: J oyce Maria: J oyce Maria Annichino Annichino--BizzachiBizzachi
HEMOCENTRO
UNICAMP
ESTUDO DA EXPRESSAO GENICAGLOBAL ENVOLVIDA NA PROTECAO
CARDIACA INDUZIDA PORPRECONDICIONAMENTO
ISQUEMICO E PORCOMPOSTOS QUINAZOLINICOS
Aluna : Ana Carolina Deckmann *Orientador: Prof. Dr. Kleber Gomes Franchini
* Laboratório de Genômica e Expressão – Departamento de Genética e Evolução – IB/ Unicamp Departamento de Clínica Médica – Faculdade de Ciências Médicas – Unicamp;

LGE

LGE

GeneProjects - interface microarrays

GeneProjects - interface microarrays

GeneProjects - interface microarrays

GeneProjects - interface microarrays

GeneProjects - interface microarrays

GeneProjects - interface microarrays

GeneProjects - interface microarrays

Perspectivas LGE
Implementar ferramenta que faça o caminho inverso.
Consolidar procedimentos para produção de chips para screening e chips para investigação direcionada.
Definir protocolos alternativos para marcação das sondas.
Implementar sistema de Facility.