Segmentação de Imagens de Cromossomos Aplicado na ... · Orientador: Valmir Macário Filho Recife...
Transcript of Segmentação de Imagens de Cromossomos Aplicado na ... · Orientador: Valmir Macário Filho Recife...

UNIVERSIDADE FEDERAL RURAL DE PERNAMBUCO
DEPARTAMENTO DE ESTATÍSTICA E INFORMÁTICA
ANGÉLICA LOPES DE JESUS
Segmentação de Imagens de Cromossomos Aplicado
na Dosimetria Biológica
TRABALHO DE CONCLUSÃO DE CURSO
Recife
Março/2017

ANGÉLICA LOPES DE JESUS
Segmentação de Imagens de Cromossomos Aplicado
na Dosimetria Biológica
Monografia apresentada ao curso de Bacha-relado em Ciências da Computação, comoparte dos requisitos necessários à obten-ção do título de Bacharel em Ciências daComputação.
Orientador: Valmir Macário Filho
Recife
Março/2017


Porque dEle e por Ele, e para Ele, são todas as coisas..
Romanos 12:36

Agradecimentos
Em ordem cronológica, agradeço primeiramente a minha vó Alvina Barbosa eminha tia Angela Gessy que foram as pessoas que me ensinaram toda a base e derammais que conhecimento e educação. Aos meus pais que sempre se esforçaram paradar o melhor pra mim desde criança e me apoiaram bastante durante o curso e a minhairmã que me ajudou na preparação das imagens para validação dos algoritmos.
A todos os professores de BCC que me ensinaram, em sua maioria muito atenci-osos e prestativos. Em especial ao professor Gilberto Cysneiros, que foi o meu primeiroorientador e que me deu a primeira oportunidade na área de iniciação cíentifica. Aoprofessor Kellyton Brito, segundo orientador, o qual ainda presta serviços de consultoriagratuita. Ao professor e orientador deste trabalho, Valmir Macário muito atenciosotambém e que durante a execução do trabalho sempre me lembrou que o tempo é curto.Aos Amigos mais próximos, Alison Andrade, Alessandro Nazário e Edcley Silva quetornaram as aulas mais agradáveis e descontraídas além das parcerias em trabalhos eprojetos.
Ao pessoal do laboratório do CRCN-NE em especial Fabiana e Mariana que mereceberam muito bem, além de esclarecer todas as dúvidas biológicas que tive durantea escrita e ajuda na rotulagem das imagens.
E por último, mas não menos importante, ao único Deus dono de toda ciênciasabedoria e poder que dá força ao cansado, e aumenta as forças ao que não temnenhum vigor.

“Dificuldades preparam pessoas comuns paradestinos extraordinários.”
Clives Staples Lewis

Resumo
Cromossomos são estruturas formadas por filamentos de moléculas de ácido deso-xirribonucleico (DNA) e várias proteínas básicas. Eles carregam toda a informaçãoque as células necessitam para seu crescimento, desenvolvimento e reprodução, re-fletindo nas características, estado de saúde e desenvolvimento do corpo. Em suavasta aplicação, a análise cromossômica é uma importante ferramenta na estimativa dadose absorvida por indivíduos expostos à radiação ionizante (dosimetria). No caso deexposição excessiva a essas radiações, é importante uma avaliação rápida e precisada estimativa dessa dose para definir o tratamento mais eficiente. Entretanto, é umprocedimento trabalhoso, lento e os sistemas automatizados são escassos e custosos.Desta forma, o objetivo desse trabalho é desenvolver um sistema para segmentar eanalisar imagens de cromossomos utilizando técnicas de visão computacional, visandoautomatizar a análise cromossômica a baixos custos. Nele estão sendo analisadasimagens de células que foram expostas à radiação ionizante, para efeito de dosime-tria. Para isso, será utilizada uma metodologia dividida em cinco etapas: i) avaliaçãoparamétrica, em que os algoritmos de binarização parametrizados serão testados afim de encontrar os parâmetros mais apropriados para cada base; ii) segmentação,para extrair/evidenciar as regiões de interesse, nesse caso, os cromossomos; iii) pósprocessamento, para melhorar a qualidade da segmentação; iv) contagem, verificaçãode quantos objetos (possíveis cromossomos) há em cada imagem; v) verificação daqualidade dos resultados. O diferencial desse trabalho é a avaliação de algoritmos debinarização (pouco usados no contexto de cromossomos), em duas bases de imagenscom diferentes características, uma delas foi disponibilizada pelo Centro Regional de Ci-ências Nucleares do Nordeste (CRCN-NE) e a outra foi disponibilizada pelo Laboratóriode Imagiologia Biomédica (BioImLab) da universidade de Padova, na Itália, somadaselas possuem um total de 112 imagens (35 do CRCN e 77 do BioImLab). Além disso,foi utilizado um algoritmo de pós processamento e de contagem bastante simples. Nosexperimentos realizados, pôde-se observar que as métricas mais apropriadas paraavaliar a segmentação de cromossomos são o coeficiente de correlação de Matthews(MCC) e a precisão. Com base nessas métricas, os algoritmos de binarização poragrupamento foram os que obtiveram melhores resultados nas bases, principalmenteos algoritmos de Otsu (MCC de 61,7% e precisão de 45,8%), K-means (MCC de 78,8%e precisão 69%) e Riddler (MCC de 65,9% e precisão de 71,8%). Por fim, notou-setambém que a forma de aquisição de imagens tem grande influência na escolha dosalgoritmos.

Abstract
Chromosomes are structures formed by filaments of deoxyribonucleic acid (DNA)molecules and several basic proteins. They carry information that the cells need fortheir growth, development and reproduction, reflection on the characteristics, state ofhealth, and development of the body. In its broad application, chromosomal analysis isan important tool in estimating the dose absorbed by an individual exposed to ionizingradiation (dosimetry). In the case of excessive exposure to these radiations, a fastand accurate assessment of the dose is important to determine which treatment ismost effective. However, it is a laborious, time-consuming procedure and automatedsystems are scarce and costly. This way, the objective of this work is to develop a sys-tem to segment and analyze chromosome images using computer vision techniques,aiming to automate the chromosomal analysis at low costs. In it are being analyzedimages of cells that were exposed to ionizing radiation, for dosimetry effect. For this, amethodology will be used, divided into five stages: i) parametric evaluation, in which theparameterized binarization algorithms will be tested in order to find the most appropriateparameters for each base ii) segmentation, to extract the regions of interest, in thiscase, the Chromosomes iii) post processing, to improve the quality of segmentation iv)counting, checking how many objects (possible chromosomes) there are in each imagev) verification of the quality of the results. The differential of this work is the evaluationof binary algorithms (little used in the context of chromosomes), in two databases ofimages with different characteristics, one of them was made available by the RegionalCenter of Nuclear Sciences of the Northeast (CRCN-NE) and the other was madeavailable by the Laboratory of Biomedical Imaging (BioImLab) of the University ofPadova, in Italy, with a total of 112 images (35 of CRCN-NE And 77 of BioImLab). Inaddition to the use of a fairly simple post-processing and counting algorithm. In theexperiments performed, it was observed that the most appropriate metrics to evaluatethe chromosome segmentation are the Matthews correlation coefficient (MCC) andthe precision. Based on these metrics, the binarization algorithms for clustering werethe ones that obtained the best results in the bases, mainly Otsu (MCC of 61,7% andprecision of 45%), K-means (MCC of 78,8% and precision of 69%) and Riddler (MCC of65,9% and precision of 71,8%) algorithms. Lastly, It was also observed that the way theimage is acquired has a great influence on the choice of algorithms.

Lista de ilustrações
Figura 1 – Cariótipo de indivíduo do sexo masculino normal . . . . . . . . . . . 15Figura 2 – Tipos de cromossomos humanos: A. metacêntrico. B. submetacênc-
trico. C. acrocêntrico . . . . . . . . . . . . . . . . . . . . . . . . . . . 16Figura 3 – Microscópio óptico utilizado no processo manual . . . . . . . . . . . 18Figura 4 – Deleção pontual antes da replicação . . . . . . . . . . . . . . . . . 23Figura 5 – Passos para formação de anéis cêntricos . . . . . . . . . . . . . . . 23Figura 6 – Passos para formação de dicêntricos . . . . . . . . . . . . . . . . . . 23Figura 7 – Exemplo curva de calibração construída in vitro . . . . . . . . . . . 25Figura 8 – a) Representação da imagem em matriz b) Exemplo de imagem
dividida em pixels . . . . . . . . . . . . . . . . . . . . . . . . . . . . 27Figura 9 – Operação pontual unária . . . . . . . . . . . . . . . . . . . . . . . . 28Figura 10 – Vizinhança entre pixels. (a) 4-conectada (b) 8-conectada . . . . . . 29Figura 11 – Áreas correlatas da imagens . . . . . . . . . . . . . . . . . . . . . . 29Figura 12 – Esquema de cores RGB representado pelo cubo . . . . . . . . . . . 30Figura 13 – a) Imagem Original b) Imagem Binarizada T=128. . . . . . . . . . . 32Figura 14 – Exemplo no ajuste global do brilho da imagem: (a) imagem original e
respectivo histograma; (b) imagem com tons de cinza atenuados erespectivo histograma; (c) imagem com tons de cinza acentuados erespectivo histograma . . . . . . . . . . . . . . . . . . . . . . . . . . 34
Figura 15 – Operação de convolução da imagem . . . . . . . . . . . . . . . . . . 35Figura 16 – imagem original (a) e imagem resultante do filtro gaussiano, σ = 2 36Figura 17 – Morfologia de imagem binária. (a) imagem original (b) dilatação (c)
erosão . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 37Figura 18 – Exemplo abertura (a) imagem com presença de ruídos (b) imagem
pós abertura . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 38Figura 19 – Algoritmos divididos em categorias . . . . . . . . . . . . . . . . . . . 41Figura 20 – Exemplo base CRCN (a) imagem original (b) segmentação ideal (ou
imagem rotulada) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 53Figura 21 – a) Segmentação ideal (b) Imagem correspondente resultante de
algoritmo avaliado (c) Matriz de confusão . . . . . . . . . . . . . . . 54Figura 22 – Exemplo da base de imagens produzidas e fornecidas pelo CRCN-NE 56Figura 23 – Microscópio óptico com câmera acoplada utilizado para aquisição
das imagens . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 57Figura 24 – Exemplo da base de imagem. Cromossomos em prometáfase (a) e
seu respectivo cariograma (b) . . . . . . . . . . . . . . . . . . . . . . 57Figura 25 – Exemplo de cromossomo com buracos ou ilhas . . . . . . . . . . . . 62

Figura 26 – Avaliação de parâmetros no algoritmo de Bernsen CRCN . . . . . . 63Figura 27 – Avaliação de parâmetros no algoritmo de Bernsen BioImLab . . . . 64Figura 28 – Avaliação de parâmetros no algoritmo de Niblack - CRCN . . . . . . 65Figura 29 – Avaliação de parâmetros no algoritmo de Niblack - BioImLab . . . . 65Figura 30 – Avaliação de parâmetros no algoritmo de Sauvola - CRCN . . . . . 66Figura 31 – Avaliação de parâmetros no algoritmo de Sauvola - BioImLab . . . . 67Figura 32 – Avaliação de parâmetros no algoritmo de K-means - CRCN . . . . . 67Figura 33 – Avaliação de parâmetros no algoritmo de K-means - BioImLab . . . 68Figura 34 – Exemplo representatividade da acurácia (a) imagem rotulada (ideal)
(b) imagem binarizada com K-means - 98, 97% (c) imagem binarizadacom Sauvola - 47,78% (d) imagem binarizada com White - 98, 52% 69
Figura 35 – Análise Sensibilidade (a) Imagem original (b) imagem binarizada peloalgoritmo Two peaks - 79,28% (c) imagem binarizada pelo algoritmoJohannsen - 0% (d) imagem binarizada pelo algoritmo Pun 100% . 70
Figura 36 – Exemplo de precisão. (a) imagem ideal (b) imagem resultante do al-goritmo Seleção iterativa 96,82% (c) imagem resultante do algoritmoPun 3,28% (d) imagem resultante do algoritmo mean 98,13% . . . . 71
Figura 37 – Representatividade MCC. (a) segmentação ideal (b) imagem resul-tante do algoritmo Seleção iterativa - 86,54% (c) imagem resultantedo algoritmo Bernsen - 59,6% (d) imagem resultante do algoritmoTwo peaks - 22,69% . . . . . . . . . . . . . . . . . . . . . . . . . . . 72
Figura 38 – Comparação da relação algoritmo, base e métricas . . . . . . . . . 73Figura 39 – Gráfico comparativo entre segmentação de Otsu e Otsu + pós pro-
cessamento . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 77Figura 40 – Exemplo da imagem após etapa de segmentação (a) e depois do pós
processamento (b) . . . . . . . . . . . . . . . . . . . . . . . . . . . . 78Figura 41 – Relação etapas x métricas - CRCN . . . . . . . . . . . . . . . . . . . 78Figura 42 – Gráfico comparativo entre segmentação de k-means e k-means +
contagem - BioImLab . . . . . . . . . . . . . . . . . . . . . . . . . . 79Figura 43 – Gráficos de dispersão dos valores resultantes e dos valores ideais
(a) base CRCN (b) base BioImLab . . . . . . . . . . . . . . . . . . . 80Figura 44 – Resultado das etapas CRCN (a) imagem original (b) imagem após
binarização com algoritmo de Otsu (c) imagem após pós processa-mento (d) após contagem . . . . . . . . . . . . . . . . . . . . . . . . 81
Figura 45 – Resultados de etapas BioImLab (a) imagem original (b) imagembinarizada com algoritmo K-means (c) imagem após contagem . . . 82

Figura 46 – Imagem da base do CRCN e MCC correspondente. Segmentaçãoideal (a) Otsu - 65,2% (b) Riddler - 64,2% (c) K-means - 70,9%(d) Seleção iterativa - 62,2% (e) Kapur - 70,4% (f) Pun - 11,6% (g)Jonhannsen - 0% (h) Sahoo - 63,3% (i) Two Peaks - 1% (j) Mean- 63,3% (k) Percentage of black - 35,5% (l) Bernsen - 60,3% (m)Niblack - 16,1% (n) Sauvola - 9,5% (o) White - 4,6% (p) . . . . . . . 89
Figura 47 – Imagem da base do BioImLab e MCC correspondente. Segmentaçãoideal (a) Otsu - 11% (b) Riddler - 79,6% (c) K-means - 88% (d) Sele-ção iterativa - 66,5% (e) Kapur - 89,3% (f) Pun - 11% (g) Jonhannsen- 99,9% (h) Sahoo - 74,6% (i) Two Peaks - 88% (j) Mean - 62,5% (k)Percentage of black - 11% (l) Bernsen - 16,5% (m) Niblack - 11,8%(n) Sauvola - 42% (o) White - 12,7% (p) . . . . . . . . . . . . . . . . 90

Lista de tabelas
Tabela 1 – Média (desvio padrão) dos resultados de cada algoritmo para cadamétrica - base CRCN . . . . . . . . . . . . . . . . . . . . . . . . . . 74
Tabela 2 – Média (desvio padrão) dos resultados de cada algoritmo para cadamétrica - base BioImLab . . . . . . . . . . . . . . . . . . . . . . . . . 75
Tabela 3 – Tempo de execução (em segundos), para uma imagem, de cadaalgoritmo em cada base de imagens . . . . . . . . . . . . . . . . . . 76

Lista de abreviaturas e siglas
CNEN Centro Nacional Energia Nuclear
CRCN - NECentro Regional de Ciências Nucleares do Nordeste
FN False Negative
FP False Positive
Gy Gray - Unidade de Dose Absorvida
HD High Definition
IAEA International Atomic Energy Agency
LSP Linfócitos Sanguíneos Periféricos
MCC Matthews Correlation Coefficient
RBF Radial Basis Function
TN True Negative
TP True Positive

Sumário
1 INTRODUÇÃO . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 151.1 Motivação . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 171.2 Objetivo . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 201.3 Objetivos Específicos . . . . . . . . . . . . . . . . . . . . . . . . . . 211.4 Estrutura . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 21
2 ASPECTOS BIOLÓGICOS . . . . . . . . . . . . . . . . . . . . . . . 222.1 Aberrações cromossômicas estruturais instáveis . . . . . . . . . 222.2 Dosimetria . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 24
3 TÉCNICAS DE PROCESSAMENTO DE IMAGENS . . . . . . . . . . 273.1 Conceitos gerais . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 273.2 Sistemas de cores . . . . . . . . . . . . . . . . . . . . . . . . . . . . 303.3 Segmentação . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 313.3.1 Binarização . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 313.4 Histograma . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 333.5 Filtros . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 353.5.1 Filtro Gaussiano . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 363.6 Operações morfológicas . . . . . . . . . . . . . . . . . . . . . . . . 373.7 Cálculos estatísticos . . . . . . . . . . . . . . . . . . . . . . . . . . 38
4 BINARIZAÇÃO DE IMAGENS DE CROMOSSOMOS . . . . . . . . . 414.1 Algoritmos de agrupamento (clustering) . . . . . . . . . . . . . . . 424.1.1 Otsu . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 424.1.2 Riddler . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 434.1.3 K-means . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 434.1.4 Seleção iterativa . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 434.2 Algoritmos de binarização baseados em entropia . . . . . . . . . 444.2.1 Kapur . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 444.2.2 Pun . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 454.2.3 Johannsen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 454.2.4 Sahoo . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 464.3 Algoritmos baseados na forma do histograma . . . . . . . . . . . 484.3.1 Two peaks . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 484.3.2 Mean gray level . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 494.3.3 Percentage of black . . . . . . . . . . . . . . . . . . . . . . . . . . . . 49

4.4 Características locais . . . . . . . . . . . . . . . . . . . . . . . . . . 494.4.1 Bernsen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 494.4.2 Niblack . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 504.4.3 Sauvola . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 504.4.4 White . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 51
5 MATERIAIS E METODOLOGIA DOS EXPERIMENTOS . . . . . . . 525.1 Verificação dos resultados . . . . . . . . . . . . . . . . . . . . . . . 525.1.1 Métricas avaliativas . . . . . . . . . . . . . . . . . . . . . . . . . . . . 535.1.2 Aquisições das imagens . . . . . . . . . . . . . . . . . . . . . . . . . 555.2 Avaliação paramétrica . . . . . . . . . . . . . . . . . . . . . . . . . . 585.3 Segmentação . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 585.4 Pós processamento . . . . . . . . . . . . . . . . . . . . . . . . . . . 595.5 Contagem . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 60
6 VALIDAÇÃO DE EXPERIMENTOS (RESULTADOS) . . . . . . . . . 636.1 Avaliação paramétrica . . . . . . . . . . . . . . . . . . . . . . . . . . 636.2 Segmentação . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 686.3 Pós processamento . . . . . . . . . . . . . . . . . . . . . . . . . . . 766.4 Contagem . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 78
7 CONCLUSÕES E TRABALHOS FUTUROS . . . . . . . . . . . . . . 83
Referências . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 84
APÊNDICES 88
APÊNDICE A – EXEMPLOS DE IMAGENS RESULTANTES DE CADAALGORITMO AVALIADO NA BASE DE IMAGENSFORNECIDA PELO CRCN-NE. . . . . . . . . . . . 89
APÊNDICE B – EXEMPLOS DE IMAGENS RESULTANTES DE CADAALGORITMO AVALIADO NA BASE DE IMAGENSDISPONIBILIZADA PELO BIOIMLAB. . . . . . . . 90

15
1 Introdução
Presentes em todas as células nucleadas de todos os organismos vivos, cro-mossomos são estruturas formadas por filamentos de um complexo de moléculas deácido desoxirribonucleico (DNA) e várias proteínas básicas. Encontrados nos núcleosdas células, eles se apresentam bem condensados durante a divisão celular, quando setornam visíveis ao microscópio óptico. Os cromossomos são responsáveis por carregartoda a informação que as células necessitam para seu crescimento, desenvolvimentoe reprodução, refletindo nas características, estado de saúde e desenvolvimento detodo corpo humano, desde a embriogênese até a reprodução (NUSSBAUM; MCINNES;WILLARD, 2008) (VERMA; BABU, 1995).
Cada espécie possui estruturas/características cromossômicas diferentes. Aestrutura cromossômica (cariótipo) humana, apresentada na Figura 1, é constituída de46 cromossomos, sendo 22 pares autossomos, identificados por números, e um par decromossomos sexuais que são identificados por letras (XX para mulheres e XY parahomens). Os pares autossomos são responsáveis por características genéticas comunsaos dois sexos; enquanto os cromossomos sexuais estão relacionados a diferentescaracterísticas dos sexos. Morfologicamente, os cromossomos humanos podem serclassificados em 3 tipos de acordo com a posição do centrômero: i. metacêntrico,quando o centrômero está localizado no meio do cromossomo; ii. submetacêntrico,quando o centrômero se encontra ligeiramente afastado do centro; iii. acrocêntrico,quando está mais próximo à extremidade, do que do centro (CUNHA, 2015) (videFigura 2).
Figura 1 – Cariótipo de indivíduo do sexo masculino normal
(Khmelinskii, 2007)

Capítulo 1. Introdução 16
Figura 2 – Tipos de cromossomos humanos: A. metacêntrico. B. submetacênctrico. C.acrocêntrico
Adaptado de Lemm (2012)
O estudo dos cromossomos, de sua estrutura e herança é chamado de Ci-togenética (NUSSBAUM; MCINNES; WILLARD, 2008). A genética médica cresceubastante no Brasil, nos últimos anos. A citogenética humana foi uma de suas primeirassubáreas a serem implantadas no país, inicialmente com laboratórios de pesquisa, emseguida, com laboratórios de análises clínicas (KURTZ et al., 2015). A Citogenética tempapel importante na medicina, pois os genes codificados no DNA têm influência sobrecaracterísticas e estado de saúde das pessoas de forma geral, tornando a análise doscromossomos um importante procedimento diagnóstico. Esta análise pode identificarvárias anomalias associadas a alterações na estrutura dos cromossomos, como asíndrome de Down (indivíduo apresenta 47 cromossomos) ou de Turner (quando háapenas um cromossomo sexual ou o segundo apresenta deformação). É essencialno diagnóstico pré-natal, permitindo o tratamento da criança ainda dentro do útero,podendo evitar que ela nasça com sérios problemas de saúde (NUSSBAUM; MCIN-NES; WILLARD, 2008).Também pode ser aplicado na identificação de vários tipos decâncer, devido a mudanças cromossômicas nas células somáticas. Além da quantifi-cação de dose de radiação ionizante absorvida por um indivíduo, que é o foco destetrabalho, através da contagem de aberrações cromossômicas (NUSSBAUM; MCINNES;WILLARD, 2008) (VERMA; BABU, 1995)

Capítulo 1. Introdução 17
1.1 Motivação
Em sua vasta aplicação, a análise cromossômica é uma importante ferramentana estimativa da dose absorvida por um indivíduo exposto à radiação ionizante (dosi-metria), seja como trabalhador, paciente ou indivíduo do público. No caso de exposiçãoexcessiva a essas radiações, é importante uma avaliação rápida e precisa da estimativadessa dose para que a equipe médica defina qual estratégia é mais eficiente no trata-mento. Entretanto, é um procedimento trabalhoso que demanda muito tempo, quandorealizado manualmente, e os sistemas automatizados são escassos e custosos (KURTZet al., 2015)(NICKOLLS et al., 1980)(SOUZA, 2011)(KURTZ et al., 2008)(SRISANG;JAROENSUTASINEE; JAROENSUTASINEE, 2006). Além disso, a experiência e habili-dade do profissinal citogeneticista é um fator importante para a correta classificaçãodas aberrações, o que torna a análise subjetiva (PANTALEÃO, 2003) .
Este trabalho é parte de um projeto desenvolvido em parceria com o CentroRegional de Ciências Nucleares do Nordeste (CRCN - NE), um instituto da ComissãoNacional de Energia Nuclear (CNEN), que atua no desenvolvimento e aplicação detecnologias nucleares e correlatas nas regiões Norte e Nordeste. O CRCN disponibilizouuma das bases de imagens de cromossomos utilizada nos testes e validações doalgoritmo, todas rotuladas por especialista, a quem será disponibilizado o sistema final.Atualmente, no CRCN, o processo de análise cromossômica é feito manualmente, ouseja, um especialista por meio de um microscópio óptico (Leica DM 500, mostrado naFigura 3) analisa ao menos 500 células em metáfase (fase do ciclo celular em que oscromossomos são visíveis) por cada amostra irradiada, contabilizando a frequênciade aberrações cromossômicas (dicêntrico, anel ou fragmento acêntrico) encontradasem cada metáfase. Para isso é necessário um tempo aproximado de 5 dias paraavaliar cada indivíduo, somado a isso um tempo de 3 a 4 dias para a preparaçãodas amostras e produção de lâminas (MATTA et al., 2013), tempo esse necessáriopara o cultivo de células. Esses aspectos motivam o uso de sistemas computacionaispara automatizar e dinamizar a análise cromossômica resultante de uma exposiçãoà radiação, especialmente necessária quando há um grande número de amostras.

Capítulo 1. Introdução 18
Figura 3 – Microscópio óptico utilizado no processo manual
Fotografado pela autora
Dentre os sistemas automatizados existentes, relacionados à dosimetria cito-genética, está o Cytoscan 110, desenvolvido em 1984 no Reino Unido, foi um dosprimeiros softwares a ser comercializado. Trata-se de um sistema de reconhecimentode imagem. Inicialmente feito para localização de metáfase, posteriormente, novasversões foram lançadas incluindo recursos de cariotipagem semi automática (FINNON;LLOYD; EDWARDS, 1986). O Medical Research Council Human Genetics (Edimburgo –Escócia) desenvolveu novas ferramentas que foram incorparadas ao sistema. Na época,esse sistema foi usado em mais de 100 laboratórios de citogenética no mundo (MATTA,2013). Outro sistema relacionado à localização de metáfase chama-se ChromoscanKaryotyping System desenvolvido pelo Cytogenetic Laboratory Clermont Ferrand, éuma aplicação voltada para computadores de uso pessoal, e foi o primeiro a utilizarredes neurais na classificação de aberrações cromossômicas. Novos equipamentosforam desenvolvidos com o passar dos anos e em 1991 foi criado o Metafer Slide Scan-ning Platform (da empresa MetaSystems® ). O sistema utiliza hardwares e softwaresmodulados, incluindo automatização de várias técnicas de biodosimetria, além de serum dos poucos sistemas que permite a detecção automática de dicêntricos (DCScoreanalysis software). Porém, além de possuir alto custo de aquisição, não é possíveladaptar seus módulos para microscopia convencional e são compatíveis apenas comMicrosoft Windows (MATTA, 2013)(METAFER. . . , ). O GeneAll HD Cariótipos1 é um1 software Geneall cariótipos acompanhado de câmera digital, cabo, adaptadores e acessórios para
captura e transmissão de imagem está avaliado em R$ 19.910,00 (dezenove mil novecentos e dezreais)

Capítulo 1. Introdução 19
software comercial aplicado no estudo do cariótipo que oferece captura de imagensem HD (High Definition), processamento de metáfases, segmentação cromossômica egeração automática de cariótipo. A ferramenta também permite que o usuário manipuleos resultados da segmentação e classificação, caso não seja considerado correto pelocitogeneticista (LABORSUL, )(KURTZ et al., 2015).
Dentre os trabalhos acadêmicos relacionados à automatização da análise decromossomos está o artigo de Kurtz et al. (2008). Eles aplicaram técnicas de visãocomputacional como suavização, realce de contraste, dilatação e limiarização parasegmentar os cromossomos, visando auxiliar o geneticista na análise da forma docromossomo, não abrangendo o tratamento de imagens com cromossomos tortos ousobrepostos. Em 2012, Poletti et al. (2012) fizeram uma comparação (com imagens seg-mentadas manualmente) entre uma variedade de métodos de thresholding/limiarizaçãovoltados para segmentação de imagens de cromossomos, concluindo que métodosque possuem limiarização local obtêm melhor desempenho, por se adaptarem melhora variações do fundo da imagem. No trabalho de MATTA (2013), foi desenvolvidauma ferramenta computacional, denominada ChromoSomeClassification, que auxilia naquantificação da dose de radiação absorvida por um indivíduo, através da contagemde aberrações cromossômicas (dicêntricos), utilizando uma rede neural RBF (RadialBasis Function) para identificá-los. Porém, ele também não trata imagens de cromos-somos sobrepostos, sendo, em alguns casos necessária a separação manual. Em2015, Kurtz et al. (2015) criaram um software que, igualmente ao primeiro citado,auxilie ao geneticista e não o substitua, no processo de elaboração do cariótipo. Elesfocaram no desenvolvimento de um software que permite ao usuário segmentar aimagem e identificar os cromossomos utilizando correlação de padrões de banda, masapesar do software identificar os cromossomos sobrepostos é necessário que o usuárioauxilie/intervenha na separação dos mesmos.Dos trabalhos relacionados, que focaram na detecção e/ou separação de cromossomoscolados e/ou sobrepostos está o de SRISANG, JAROENSUTASINEE e JAROENSU-TASINEE (2006), em que foi desenvolvida uma técnica para separar cromossomossobrepostos baseada na geometria computacional (diagrama de Voronoi e triangulaçãode Delaunay) alcançando altas taxas de acerto. Grisan, Poletti e Ruggeri (2009) tam-bém criaram uma metodologia de segmentação de cromossomos que se tocam e/ouse sobrepõem gerando, para cada conjunto de cromossomos sobrepostos, uma árvorecom possíveis formas para desembaraçá-los. A metodologia foi avaliada em imagensobtidas na prometáfase, que foram misturados na quinacrina para permitir que os cro-mossomos se tornem visíveis (Q-band). Minaee, Fotouhi e Khalaj (2014) utilizaramcritérios geométricos para identificar os cromossomos que se tocam e se sobrepõemparcialmente encontrando uma linha de corte e separando-os, alcançando bons resul-tados. Madian, Jayanthi e Dr.S.Suresh (2015) propõem um algoritmo para identificar

Capítulo 1. Introdução 20
a zona de sobreposição de cromossomos na imagem sem nenhuma intervenção hu-mana, utilizando funções de curvatura para identificar os pontos de corte e realizar aseparação, conseguindo fazê-la com uma acurácia de 96% de acerto, sugerindo seuuso como etapa anterior à segmentação.Em relação à classificação de cromossomos, M. e Emary (2006) desenvolveram umfiltro para remoção de ruídos melhorando a qualidade da imagem em um fator iguala 91,7% em seguida classificando-os pelo tamanho; utilizando a técnica de esque-letização e utilizando uma rede neural com o algoritmo backpropagation em que asimagens segmentadas são as entradas da rede. Khmelinskii (2007), em sua dissertaçãode mestrado, desenvolveu 4 classificadores voltados para emparelhamento de cromos-somos para propósito de cariotipagem, trabalhando numa base de imagens metafásicas,já segmentadas, manualmente. Poletti, Grisan e Ruggeri (2008) desenvolveram um sis-tema de classificação de cromossomos robusto, utilizando pré-processamento e redesneurais numa base de imagens contendo 119 cariótipos (forma gráfica de organizaros cromossomos do maior para o menor, diferenciando por grupos), segmentadosmanualmente e rotulados por um especialista, obtendo acurácia de 95,6%. Kurtz (2011),em sua dissertação de mestrado, propôs métodos para detecção de centrômeros doscromossomos, definindo dois algoritmos baseados nesses métodos. Alcançando taxasde acerto de até 94%, o autor sugere a detecção do centrômero como etapa anterior àsegmentação e identificação de cromossomos.
Neste trabalho foram avaliados 15 algoritmos de binarização. Muitos deles fo-ram poucas vezes (ou nenhuma vez) aplicados no contexto de cromossomos, como:Bernsen, Niblack, Sauvola, White, Percentage of black, Pun, Sahoo, Kapur, Johann-sen, Mean gray level, Two peaks e Seleção iterativa. Todos eles foram avaliadosdiretamente, isto é, sem a aplicação de pré-processamento. Foram utilizadas quatrométricas (MCC, precisão, sensibilidade e acurácia) em duas bases de imagens comdiferentes características quanto a presença de ruídos, iluminação e contraste. Fo-ram desenvolvidos também, algoritmos simples de pós processamento e contagem queobtiveram um aumento de até 10% no valor do MCC, principal métrica usada no critériode classificação dos algoritmos. Não é escopo desse trabalho a detecção e separaçãodos cromossomos colados e sobrepostos nem a classificação dos cromossomos quantoao tipo de aberração cromossômica.
1.2 Objetivo
Desenvolver um sistema para segmentar e analisar imagens de cromossomosutilizando algoritmos de binarização automática.

Capítulo 1. Introdução 21
1.3 Objetivos Específicos
1) Automatizar o processo de análise cromossômica utilizando algoritmos de binari-zação automática, visando agilizar o processo.
2) Segmentar os cromossomos de uma dada imagem, para possibilitar, posteri-ormente em trabalhos futuros, identificá-los e classificá-los quanto ao tipo deaberração estrutural cromossômica.
3) Aplicar técnicas de pós-processamento de imagens para melhorar a qualidade daimagem a ser analisada.
4) Contar a quantidade de cromossomos contidos nas imagens, permitindo identificarse existe algum tipo de alteração do número de cromossomos.
1.4 Estrutura
O capítulo 2 apresentará conceitos biológicos, no contexto do trabalho, relaci-onados à dosimetria e aberrações cromossômicas. O capítulo 3 introduz as técnicasprocessamento de imagens abordadas neste trabalho, como segmentação, histogramae filtros. O capítulo 4 apresenta o conceito e utilização de algoritmos de binarizaçãoutilizados na segmentação dos cromossomos. O capítulo 5 apresenta os materias ea metodologia utilizada nos experimentos e validação dos resultados. O capítulo 6descreve os resultados obtidos em cada etapa dos experimentos. E no capítulo 7, aconclusão e os trabalhos futuros.

22
2 Aspectos Biológicos
Quando os indivíduos são expostos à radiação ionizante natural ou artificial,seus cromossomos podem sofrer alterações morfológicas, o que indica que há danosno material genético ou DNA, (SOUZA, 2011) essas alterações são comumente cha-madas de aberrações ou mutações cromossômicas. Quando a alteração está ligada aoaumento ou diminuição do número de cromossomos denomina-se mutação numérica.Quando está relacionada ao rearranjo dos genes nos cromossomos, geralmente alte-rando a morfologia do cromossomo, são chamadas de mutações estruturais. Ambas asalterações (estruturais ou numéricas) ocorrem espontaneamente por fatores internosou externos à célula (CUNHA, 2015) e resultam em anomalias que podem causardesequilíbrios na formação, no crescimento, no desenvolvimento e no metabolismo dascélulas.
Com relação à fase do ciclo celular onde ocorre a ação dos agentes genotóxicos,as aberrações por radiação induzida podem ser classificadas basicamente de duasformas: do tipo cromossômico e do tipo cromatídico. Nas do tipo cromossômico, osagentes genotóxicos atuam antes da duplicação do DNA e nas do tipo cromatídicoatuam após a duplicação do DNA e envolvem apenas uma das cromátides (filamento deDNA) do cromossomo (SOUZA, 2011). As aberrações do tipo cromossômica estruturalserão as observadas neste trabalho, por isso serão abordadas com mais detalhes aseguir.
2.1 Aberrações cromossômicas estruturais instáveis
Dentre as alterações cromossômicas instáveis mais comuns, estão: os fragmen-tos isolados, os anéis, e os dicêntricos (MENDES, 2014). As alterações chamadasde fragmentos isolados podem ser divididas em: I) Fragmentos acêntricos, tambémchamados de deleção terminal, em que o cromossomo é rompido e perde partes(fragmentos) de ambas as cromátides. Eles encontram-se paralelos entre si, sem ne-nhum centrômero entre eles. II) Deleção pontual ou intersticial, ocorre quando há duasquebras no mesmo braço do cromossomo onde o fragmento da extremidade se une aorestante do cromossomo deixando o fragmento intermediário livre, como mostrado naFigura 4.

Capítulo 2. Aspectos Biológicos 23
Figura 4 – Deleção pontual antes da replicação
(Hall;Giaccia, 2006)
A mutação estrutural denominada Anel ocorre quando uma cromátide comquebra nas duas extremidades, unem essas novas extremidades em formato de anelpodendo ser acêntrico (sem centrômero) ou cêntrico (quando há um centrômero). AFigura 5 mostra como são formados os aneis cêntricos.
Figura 5 – Passos para formação de anéis cêntricos
Adaptado de Hall;Giaccia(2006)
Inicialmente ocorre quebra nos dois braços de um mesmo cromossomo (II) emseguida as extremidades ligadas ao centrômero se unem incorretamente em forma deanel e as demais partes se unem formando um fragmento acêntrico (III) então ocorre areplicação resultando em dois círculos ligados pelo mesmo centrômero.
Os cromossomos dicêntricos, também chamados de translocações assimétricasou policêntricas mais complexas, são considerados o “padrão ouro” da biodosimetria eocorrem quando dois cromossomos distintos se rompem e se reintegram incorreta-mente, havendo troca de material, e formando uma estrutura com dois centrômeros.A Figura 6 mostra como é formado esse tipo de aberração. Quando mais de doiscromossomos são rompidos, no caso de exposição à radiação ionizante resultandoem altas doses absorvidas, estruturas policêntricas são produzidas.
Figura 6 – Passos para formação de dicêntricos
Adaptado de Hall; Giaccia(2006)

Capítulo 2. Aspectos Biológicos 24
A primeira imagem (I) da Figura 6 mostra dois cromossomos distintos antesda replicação, cada um desses cromossomos sofre uma ruptura (II) no início daintérfase, como as extremidades coesivas estão próximas, elas se unem de formainvertida/ilegítima (III) dessa forma, trocando material genético. Em seguida, (IV) essaaberração se replica durante a síntese de DNA gerando um cromossomo com doiscentrômeros (dicêntricos), além de um fragmento sem nenhum centrômero (fragmentoacêntrico).
2.2 Dosimetria
Dosimetria é uma medição criteriosa relacionada às grandezas radiológicaspara fins de controle, registro e proteção de indivíduos ocupacionalmente expostose pacientes submetidos a práticas que envolvem o uso de radiação ionizantes (DEL-GADO, 2000). Quanto à forma de exposição, ela pode ser classificada como externa -quando a fonte é externa ao indivíduo - ou interna quando há incorporação do materialradioativo (CRCN-NE, 1996). Quanto à forma de se estimar a dose pode-se adotarmétodos físicos (dosimetria física), ou seja com auxílios de dosímetros, métodos com-putacionais (simulação) ou métodos biológicos. Os métodos biológicos, que compõema dosimetria biológica, foram criados para confirmar a dosimetria física ou para seremusados quando outras medidas, incluindo a física, não estão disponíveis no momentoda exposição. A dosimetria biológica utiliza indicadores biológicos (biomarcadores)sensíveis e específicos à radiação, relacionando a dose absorvida (concentração demedida de radiação por unidade de massa do volume irradiado) e os efeitos biológicoscausados pela radiação (MENDES, 2014). Os biomarcadores podem ser classificadosem 3 classes: de sensibilidade, de exposição e de doença. Biomarcadores de sensi-bilidade são marcadores genéticos relacionados com o aumento da susceptibilidade,dos danos causados pela radiação, como marcadores moleculares. Por exemplo, oaumento da expressão da proteína p53. Biomarcadores de exposição são parâmetrosbiológicos que podem ser associados a uma relação dose-resposta, como a dosimetriacitogenética que faz uso de aberrações cromossômicas e/ou micronúcleos. Biomarca-dores de doenças são manifestações clínicas, sintomas de doenças (vômitos, diarréia,radiodermatite etc), quedas de hemácias e plaquetas entre outros (MATTA, 2013).Dentre estes, a dosimetria citogenética é a mais confiável em situação de exposiçãoexcessiva à radiação (MATTA et al., 2013). Algumas técnicas usadas na dosimetria cito-genética são: análise de dicêntricos, condensação prematura dos cromossomos (PCC),micronúcleos por bloqueio da citocinese, hibridização in situ fluorescente (FISH), entreoutras.
Desde a década de 60, a técnica de análise de dicêntricos tem sido utilizadana análise de dose absorvida. Ela utiliza aberrações cromossômicas formadas nos

Capítulo 2. Aspectos Biológicos 25
linfócitos sanguíneos periféricos (LSP) expostos à radiação ionizante, relacionando afrequência das aberrações encontradas com a dose absorvida tanto in vitro quanto invivo (SILVA, 1997). Os linfócitos sanguíneos são células de defesa responsáveis poreliminar agentes infeciosos que entram em contato com o organismo, dando imunidadeao corpo. Eles são utilizados por serem reconhecidos como células altamente sensíveisà radiação ionizante. Isso significa que o tempo entre a irradiação e a manifestaçãodo efeito é bem menor nos linfócitos periféricos (MENDES, 2014). Outra vantagem nautilização dos linfócitos é a baixa frequência de dicêntricos encontrados na populaçãosaudável - cerca de um dicêntrico em 1000 células (AINSBURY et al., 2011).
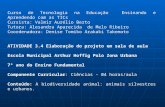
A quantidade de aberrações cromossômicas encontradas nos linfócitos são con-vertidas em doses absorvidas de acordo com uma curva de calibração dose-resposta,previamente estabelecida. As curvas de calibração dose-resposta foram desenvol-vidas para representar matematicamente a relação entre frequência de aberraçõesencontradas nos linfócitos e a dose absorvida pelo organismo (MENDES, 2014). Essacurva referencial é obtida através de irradiação de amostra de sangue de um doadorvoluntário saudável, e só é possível sua utilização porque ao irradiar linfócitos in vitrode forma similar a situação in vivo se obtem a mesma relação dose-resposta (MATTA,2013). A Figura 7 mostra exemplos de curvas de calibração construída in vitro, nelaforam relacionadas a quantidade de dicêntricos por célula e a dose absorvida dada emGy para diferentes tipos de radiação com diferentes formas de dose absorvida.
Figura 7 – Exemplo curva de calibração construída in vitro
(Lloyd DC;Purrott, 1981)
Cada ponto da curva representa uma média da dose de radiação absorvida (eixo

Capítulo 2. Aspectos Biológicos 26
das abscissas) e a frequência de aberrações (na imagem, apenas dicêntricos) contidosnos linfócitos irradiados e coletados. Essa dose é equivalente a dose de corpo inteiro,dado que os linfócitos são bastante móveis e distribuídos pelo corpo. É recomendadopela IAEA (International Atomic Energy Agency) que cada laboratório construa suaprópria curva de calibração, pois a utilização de curvas construídas em outros lugarespode trazer mais incertezas à estimativa da dose absorvida (MENDES, 2014).
O algoritmo e análise estudadas e desenvolvidas nesse trabalho, que serão des-critos nos próximos capítulos, compreendem as primeiras etapas necessárias/essenci-ais para o objetivo final que é encontrar a frequência de cada aberração cromossômica.Após encontrar essas frequências a dose será determinada pela relação construída nacurva de calibração descrita acima.

27
3 Técnicas de processamento de imagens
3.1 Conceitos gerais
Imagens são representações visuais de objetos ou cenas físicas, que podemser armazenadas, manipuladas e interpretadas conforme aplicação de estudo. Mate-maticamente, ela pode ser descrita como uma função de intensidade luminosa refletidado objeto, considerando, geralmente, o espaço bidimensional. Ela pode ser definidacomo f(x, y), onde x e y são as coordenadas no espaço e o valor de f na coorde-nada (x, y) refere-se ao valor/intensidade, ou seja, o brilho da imagem no ponto.
Imagens podem ser classificadas como analógicas ou digitais. As imagensdigitais são representadas por matrizes de valores discretos. Para digitalizar umaimagem analógica é necessário que ela seja discretizada e para isso são utilizados doisprocessos denominados de amostragem e quantização. A amostragem é responsávelpela discretização das coordenadas espaciais e após esse processo, a imagem édividida em pequenos pontos/quadrados. A quantização é a discretização da quantidadede brilho de cada ponto. O valor assumido por cada ponto diz respeito à quantificação,dada por um número definido em escala referente ao sistema de cor da imagemrepresentada, por exemplo, se a imagem representada estiver na escala de cinzaesse número/valor estará numa faixa de valores entre 0 e 255. Cada ponto da matriz-imagem produzida é chamado de “Pixel” uma abreviação do inglês Picture Element. Umpixel é a menor unidade discernível em uma imagem, sobre o qual podemos realizaroperações. O tamanho de um pixel está relacionado com a resolução espacial com aqual foi adquirida. A representação matemática da imagem possibilita a manipulação etranformação do seu conteúdo, a fim de evidenciar ou remover informações da mesma.Às várias técnicas e operações que se pode aplicar a uma matriz-imagem dá-se onome de processamento de imagens (NUNES, 2006). A Figura 8 mostra a forma derepresentar a imagem matematicamente (utilizando matriz) e um exemplo de imagemdividida em pixels.
Figura 8 – a) Representação da imagem em matriz b) Exemplo de imagem dividida em pixels
Adaptado de Nunes (2006)

Capítulo 3. Técnicas de processamento de imagens 28
Dentre os vários tipos de operações que podem ser realizadas sobre imagens,neste trabalho serão utilizadas as denominadas operações no domínio espacial. Essasoperações se caracterizam pela manipulação direta dos pixels. Uma operação genéricaOp sobre uma sequência de n imagens, I1, I2, I3...In obtendo como resultado umaimagem IS pode ser representada como
IS = Op(I1, I2, I3..In) (3.1)
As operações Op podem ser qualquer operação aritmética, lógica, de compa-ração, entre outras. Essas operações são denominadas n-árias, pois a imagem desaída resulta da combinação de duas ou mais imagens de entrada. Com relação aoescopo, as operações do domínio espacial podem ser classificadas como pontuais(ponto-a-ponto) ou locais (localizadas). Nas operações pontuais, cada pixel da imagemde saída ps(x, y) depende unicamente do pixel da imagem original de localizaçãocorrespondente po(x, y), podendo ser interpretadas como um mapeamento de pixelsda imagem de entrada para a imagem de saída, como mostra a Figura 9 (QUEIROZ;GOMES, 2001).
Figura 9 – Operação pontual unária
(Queiroz; Gomes, 2001)
Por outro lado, nas operações denominadas locais, os valores de um pixel dedeterminada coordenada (x, y) dependem do valor de entrada dessa coordenada e dasua vizinhança. Os tipos de vizinhança mais comumente utilizadas são as chamadas 4-conectada e 8-conectada. Os vizinhos mais próximos, dado um pixel p com localização(i, j) são os pixels de coordenadas (i+ 1, j), (i− 1, j), (i, j + 1) e (i, j − 1) essavizinhança é denominada 4-conectada. Os vizinhos mais distantes que possuem ascoordenadas (i− 1, j− 1), (i− 1, j + 1), (i+ 1, j− 1) e (i+ 1, j + 1) juntamentecom os vizinhos mais próximos formam a vizinhança chamada 8-conectada. A Figura10 mostra a relação entre os pixels.

Capítulo 3. Técnicas de processamento de imagens 29
Figura 10 – Vizinhança entre pixels. (a) 4-conectada (b) 8-conectada
Elaborado pela autora
Dentre as áreas da computação que estão relacionadas a imagens, o proces-samento de imagens é utilizado para melhorar a qualidade da imagem e/ou realçarcaracterísticas/informações contidas nela (BALLARD; BROWN, 1982). Trata-se datransformação de uma imagem em outra imagem. Difere da visão computacional, queextrai informações da imagem gerando uma descrição explícita dos objetos contidosnela, tornando capaz a interpretação da imagem por um sistema computacional. Jáa computação gráfica refere-se à síntese de imagens ou geração de imagens a partirda descrição, de um modelo. A Figura 11 mostra a relação entre essas áreas.
Figura 11 – Áreas correlatas da imagens

Capítulo 3. Técnicas de processamento de imagens 30
3.2 Sistemas de cores
A luz é uma onda eletromagnética que se propaga através de diferentesmeios. Sendo uma onda, ela pode ser caracterizada pela sua frequência (f ) e pelo seucomprimento de onda (λ). A faixa de espectro de luz percebido pelo olho humano édefinida num intervalo de, aproximadamente 400 a 700nm e é denominada luz visível.Dentro desse intervalo o olho pode perceber várias cores (ou comprimentos de onda)diferentes, mas quando há emissão de apenas um comprimento de onda, ou seja,apenas uma cor pode ser notada, a luz é denominada monocromática (QUEIROZ;GOMES, 2001). As cores dos objetos são dadas pela reflexão dos comprimentos deonda que não foram absorvidos por eles, sendo então refletidos, tornando-se visíveis aoolho humano.
Os sistemas de cores, também chamados de espaço de cores ou modelo decores, foram desenvolvidos para padronizar a especificação das cores. Trata-se basica-mente de um sistema de coordenada ou subespaço em que cada cor é representadapor um único ponto (GONZALEZ; WOODS, 2007). O sistema de cores mais comu-mente usado, em termos de processamento digital, é o RGB (Red, Green e Blue).Aplicado nas cores dos monitores e em câmeras de vídeo, o RGB utiliza três canais,um para cada componente espectral primário (verde, azul e vermelho) e cada canal érepresentado utilizando 8 bits. Sendo assim, cada pixel da imagem colorida, no sistemaRGB, pode assumir (28)3 = 224 = 16,777,216 possíveis cores. Em uma imagem comtamanho M x N a quantidade de bits utilizados para representar uma imagem em RGBé dada por M x N X (lR +lG + lB) sendo lR, lG e lB a quantidade de bits utilizada emcada canal. O modelo RGB é baseado no sistema de coordenadas cartesiano e temseu subespaço representado pelo cubo de cores mostrado na Figura 12.
Figura 12 – Esquema de cores RGB representado pelo cubo
Adaptado de Queiroz; Gomes(2001)

Capítulo 3. Técnicas de processamento de imagens 31
Todos os valores de R, G e B estão num intervalo de [0,1]. Nos vértices do cuboestão as cores primárias (vermelho, verde e azul) a combinação delas, duas a duas,utilizando a mesma intensidade, gera as chamadas cores secundárias (ciano, magentae amarelo), também representadas no cubo. O preto está localizado na origem do planocartesiano e o branco na extremidade oposta (QUEIROZ; GOMES, 2001) (GONZALEZ;WOODS, 2007). A escala de cinza fica representada na diagonal entre as extremidadespreta e branca. Um procedimento comum no processamento digital de imagens é aconversão de imagens coloridas (RGB) para imagens em tons de cinza (escala decinza). Isso ocorre pelo fato de imagens em tons de cinza precisarem de apenas umcanal para sua representação sendo mais fácil a sua manipulação, já que cada pixel érepresentado por apenas um número (valor de intensidade). A faixa de valores que umpixel pode assumir na escala de cinza compreende o intervalo de 0 a 255.
3.3 Segmentação
Segundo Gonzalez e Woods (2007), segmentação consiste em subdividir umaimagem em parte ou objetos constituintes. Ela possibilita a identificação e/ou distinçãoentre dois ou mais objetos entre si e o background (fundo da imagem). Trata-se de umagrupamento de partes de uma imagem generalizada em uma imagem homogêneaconsiderando características da imagem. A abordagem mais comum de segmentaçãochama-se limiarização (thresholding), ou binarização, descrita na Subseção a seguir.
3.3.1 Binarização
Binarização, ou limiarização, é o processo de conversão de uma imagem ori-ginalmente em tons de cinza para uma imagem binária (com dois tons), geralmentepreto e branco (os extremos da escala de cinza). Esse processo é utilizado quando emdeterminada situação é necessária a separação da região de interesse (foreground, ouprimeiro plano) do fundo da imagem (background). Para isso, ela separa os agrupa-mentos baseado em um limiar (ou Thresholding). A binarização é feita varrendo-se aimagem pixel a pixel e alterando o valor do pixel conforme relação dele com o limiar. Aimagem limiarizada g(x, y) é definida como:
g(x, y) =
0, f(x, y) ≤ T
1, f(x, y) > T(3.2)
Onde, f(x, y) denota o nível de cinza do ponto (x, y) e T o valor do limiar.Quando T depender apenas de f(x, y) o limiar será chamado de global, quando

Capítulo 3. Técnicas de processamento de imagens 32
depender de outra propriedade local do ponto (x, y) além de f(x, y), será chamadode dinâmico (BERTHOLDO, 2007).
Binarização é um dos métodos mais simples de segmentação de imagens.Quando há apenas um valor de limiar a técnica é denominada limiarização globalsimples. A maior dificuldade na aplicação da limiarização é a definição do valor do limiar,sendo uma das formas utilizadas a análise do histograma da imagem. Porém, em algunscasos, não é possível encontrar um único limiar que obtenha uma segmentação ideal.Para esses casos existem técnicas de limiarização variáveis e multiníveis baseadasem várias medidas estatísticas. A Figura 13 mostra um exemplo da aplicação daLimiarização com o valor do limiar igual a 128.
Figura 13 – a) Imagem Original b) Imagem Binarizada T=128.
Adaptado de Nunes(2006)
Há várias possíveis formas de classificar algoritmos de binarização. No estudode Sezgin e Sankur (2004), foi desenvolvido uma taxonomia voltada para esse tipo dealgoritmo, onde foram definidos seis grupos ou categorias de acordo com as informa-ções que eles exploram na imagem, são eles: i. métodos de agrupamento (clustering):a ideia principal desses métodos consiste em separar os dados (níveis de cinza), ouobjetos, em grupos de acordo com as características similares que eles possuemresultando sempre em duas classes; ii. métodos baseados em entropia: nesta categoriaa ideia é explorar a distribuição da entropia dos níveis de cinza em uma imagem; iii. mé-todos baseados na forma do histograma: nesta classe os métodos encontram o limiarcom base nas propriedades de forma do histograma (e.g., curvaturas, picos e vales);iv. métodos locais: nesta categoria é calculado um limiar para cada região da imagem,levandoem consideração apenas os pixels da vizinhança, não os da imagem toda; v. métodosbaseados nos atributos do objeto: que buscam uma medida de similaridade entre onível de cinza e a imagem binarizada; vi. métodos espaciais: que utilizam distribuiçãode probabilidade de ordem superior e/ou correlação entre pixels.

Capítulo 3. Técnicas de processamento de imagens 33
No Capítulo 4 serão descritos os metódos de binarização avaliados nessetrabalho.
3.4 Histograma
Histograma é uma descrição global, ou representação gráfica da distribuição detons de cores existente em uma imagem. Quando a imagem utiliza o sistema de coresRGB, cada canal de cor é descrito por um histograma e uma composição deles geraum histograma geral. Quando a imagem utiliza a escala de cinza, um único histogramaé suficiente para representar a distribuição de níveis de cinza da imagem. Histogramassão a base para inúmeras técnicas de processamento de imagens, ele pode ser usadopara melhorar a qualidade da imagem e prover estatísticas, além de possuir simplesimplementação em software e ter uma implementação econômica em hardware. Porém,não é ideal para aplicações que necessitem de informações da localização dos pixels,pois ele não armazena esse dado (GONZALEZ; WOODS, 2007). Um histogramapode ser expresso como uma função discreta h(rk) = nk, onde rk é o k-ésimovalor de intensidade r e nk é a quantidade de ocorrência de pixels com intensidadek na imagem. Esse valor é, normalmente, normalizado, ou seja, cada componenteda imagem é dividido pelo número total de pixels da imagem. Assim um histogramanormalizado pode ser expresso como:
P (rk) =nkMN
(3.3)
onde k = 0, 1, 2, 3, 4, ...L− 1 níveis de cinza e M e N as dimensões da matriz-imagem. P (rk) representa a distribuição de probabilidade dos pixels, isto é, a proba-bilidade de ocorrência de um nível de intensidade rk. A soma dos componentes dohistograma normalizado é sempre igual a 1. Os valores obtidos no cálculo de cadacomponente geralmente são representados num gráfico de barras com a abscissarepresentando os níveis de intensidade e o eixo da ordenadas, o percentual de pixels. Avisualização gráfica do histograma permite a rápida percepção da qualidade da imagemcom relação ao nível de contraste e brilho presentes na imagem, como mostra a Figura14.

Capítulo 3. Técnicas de processamento de imagens 34
Figura 14 – Exemplo no ajuste global do brilho da imagem: (a) imagem original e respectivohistograma; (b) imagem com tons de cinza atenuados e respectivo histograma;
(c) imagem com tons de cinza acentuados e respectivo histograma
(Nunes, 2006)

Capítulo 3. Técnicas de processamento de imagens 35
3.5 Filtros
Filtros são técnicas que permitem destacar aspectos da imagem ou reduzirruídos. Eles podem ser classificados como passa-alta (deixa passar as altas frequênciasmas elimina os valores relacionados às baixas frequências), passa-banda (os valoresdo sinal da imagem que pertecem à determinada frequência pernanecem e os demaissão eliminados) ou passa-baixa (deixa passar as baixas frequências e elimina osvalores relacionados a altas frequências).
A técnica de filtragem de imagem é basicamente uma transformação pixel apixel com base, não apenas na intensidade de determinado pixel da imagem original,mas na intensidade dos pixels da vizinhança dele. A filtragem espacial é uma operaçãolocal, que depende do contexto que cada pixel está inserido. Ela é fundamentada naoperação de convolução entre uma máscara (mask, kernel, template ou janela) e aimagem a ser filtrada. A máscara é uma matriz com dimensões inferiores a da imagemoriginal, geralmente quadrada e com valores definidos como pesos a serem aplicadossobre pixels da imagem. A operação é executada progressivamente sobre cada pixelda imagem (QUEIROZ; GOMES, 2001).
Figura 15 – Operação de convolução da imagem
Adaptado de Queiroz e Gomes (2001)
Dada a Figura 15, se o centro da máscara p5 estiver sobre o pixel da posição(x,y) da imagem, o tom do pixel da posição (x,y) será alterado para o valor resultanteda soma dos produtos dos pesos da máscara com o valor dos pixels da imagem

Capítulo 3. Técnicas de processamento de imagens 36
que esta sob filtro (ou vizinhança do pixel central). O processo de mover a máscarasobre a imagem calculando a soma dos produtos de cada pixel da imagem chama-seconvolução (GONZALEZ; WOODS, 2007).
Nas Subseções seguintes, serão descritos os filtros utilizados na etapa de pósprocessamento deste trabalho.
3.5.1 Filtro Gaussiano
O filtro gaussiano pode ser usado como filtro passa-baixa, portanto tem a funçãode “borrar”, ou suavizar (blur ) a imagem já que as altas frequências, responsáveispelas transições abruptas, são atenuadas. A suavização tende a diminuir o ruído poisos pequenos detalhes da imagem são perdidos (MÜLLER; DARONCO, 2000). O filtroGaussiano tem esse nome por utilizar a função gaussiana (mostrada na Equação 3.4)para obter os valores da máscara.
G(x, y) =1
2Πσ2e−
x2+y2
σ2 (3.4)
Na equação acima, quanto maior o desvio padrão σ maior o grau de suavização.Em duas dimensões, esse filtro possui o grau de suavização igual em ambas asdireções, pois funções gaussianas são simétricas em relação à rotação. A suavizaçãoda imagem é realizada através da substituição de cada pixel pela média ponderadados pixels vizinhos. O grau de suavização do filtro gaussiano também está relacionadoao tamanho da máscara/janela, quanto maior o tamanho da máscara, maior será ograu de suavização aplicado na imagem. A Figura 16 abaixo mostra um exemplo daaplicação do filtro gaussiano (SANCHES et al., 2015).
Figura 16 – imagem original (a) e imagem resultante do filtro gaussiano, σ = 2
Fonte: https://www.mathworks.com/help/images/ref/imgaussfilt.html

Capítulo 3. Técnicas de processamento de imagens 37
3.6 Operações morfológicas
As operações morfológicas extraem informações relativas à geometria e topo-logia dos objetos desconhecidos da imagem, através de um conjunto completamentedefinido chamado elemento estruturante. A forma e tamanho do elemento estruturantepermitem testar e quantificar de que maneira ele “está ou não está contido” na imagem(FACON, 2013). As operações morfológicas são tipicamente usadas para remoçãode imperfeições que surgem durante o processo de segmentação. A vantagem damorfologia matemática é que seus operadores baseam-se em poucos operadoresbásicos. Existem 4 operadores morfológicos básicos: erosão, dilatação, abertura efechamento.
Na prática a erosão reduz os objetos presentes em uma imagem, sendousada para eliminar pequenos ruídos ou separar objetos que estejam colados. A ero-são de uma imagem f por um elemento estruturante g é definida por erog(f(x)) =Min
{f(y) − g(x − y) : y ∈ E
}. Já a dilatação é definida por dilg(f(x)) =
Max{f(y) + g(x − y) : y ∈ E
}e, na prática, ela expande os objetos contidos
na imagem, sendo comumente usada para reparar quebras ou falhas dos itens daimagem. A Figura 17, abaixo, mostra um exemplo da aplicação dessas duas operaçõesmorfológicas.
Figura 17 – Morfologia de imagem binária. (a) imagem original (b) dilatação (c) erosão
Elaborada pela autora
As propriedades da iteração da erosão e dilatação permitem criar novos opera-dores como a abertura. A abertura de uma imagem f por um elemento estruturante gconsiste em dilatar f pelo transposto de g (g) e em seguida dilatar o conjunto erodidopor g.
abeg(f) = dilg(erog(f)) (3.5)

Capítulo 3. Técnicas de processamento de imagens 38
A operação de abertura é utilizado para remover os pequenos ruídos sem queas propriedades de forma do objeto sejam alteradas. A Figura 18, abaixo, mostra umexemplo da atuação da abertura na remoção de ruídos. Na seção 6.3 é mostrado oresultado da abertura em imagens de cromossomos.
Figura 18 – Exemplo abertura (a) imagem com presença de ruídos (b) imagem pós abertura
Adaptado de Parker (2011)
3.7 Cálculos estatísticos
Nesta Seção, serão descritas algumas medidas estatísticas utilizadas pelosmétodos de binarização e na análise dos resultados.
Dentre as métricas, a mais simples e bastante utilizada é a média aritméticasimples. A média aritmética (denotada por µ ou x) é uma medida descritiva de posição(ou de tendência central) que tem seu uso restrito para dados quantitativos. O valorde média de um conjunto de dados é obtido na divisão da soma de todos os dados doconjunto pela quantidade total de dados. No contexto de imagens, a média é obtida nadivisão da quantidade de pixels que a imagem possui com a mesma intensidade/valor,sobre o número total de pixels. Conforme Equação 3.6 onde n representa o númerototal de pixels.
x =x1 + x2 + x3 + ...xn
n=
1
nΣni=0xi (3.6)
A média representa o valor central da distribuição e pode ser interpretadacomo o centro de gravidade dos dados de maneira que se todos os dados fossemsubstituídos por ela, a soma total permaneceria igual à soma dos valores originais(FARIAS; LAURENCEL, 2006). A média é a métrica indicada quando a distribuição dosdados é simétrica em relação a um valor médio porém, quando há um espalhamento

Capítulo 3. Técnicas de processamento de imagens 39
muito grande dos dados, a média não é uma medida boa para representar/resumir osdados, pois é bastante influenciada por valores extremos do conjunto de dados. Emcasos onde existe um grande espalhamento na distribuição dos dados, medidas dedispersão como: desvio padrão e variância são mais indicados.
A variância (s2ou σ2) é a média dos desvios quadráticos, “o somatório doquadrado da distância dos valores observados em relação à média dividido pelo totalda amostra menos um” que utiliza todos os pixels da distribuição, definida pela equação3.7.
s2 =Σni=1(xi − x)2
n− 1(3.7)
Na Equação 3.7, quando relacionada a imagem, o valor de n representa aquantidade total de pixels, xi os valores dos pixels e x a média aritmética da imagem.Em termos de imagem, a variância e a média caracterizam a iluminação obtida durantea aquisição da imagem. A variância também indica a variação das intensidades de cinzaem relação a sua média, caracterizando o contraste da imagem. Uma desvantagem davariância é que a unidade de medida do valor resultante é a mesma dos dados, porémelevado ao quadrado. Uma maneira de se obter o resultado com mesma unidade dosdados consiste em obter a raiz quadrada da variância que é denominada desvio padrão.O desvio padrão (denotado por s ou σ) estima quanto cada valor do pixel está longe damédia aritmética simples, mantendo a mesma unidade de medida dos dados originais.Matematicamente é representado pela raiz quadrada da variância, conforme Equação3.8.
s =2√s2 =
2
√Σni=1(xi − x)2
n− 1(3.8)
Dentre as categorias que foram divididos os algoritmos de binarização, está acategoria que utiliza o valor da entropia dos dados. Entropia pode ser definida em váriasáreas do conhecimento (e.g., química, termodinâmica e até economia). De forma geral,está relacionada com estado de ordem ou desordem de um sistema caracterizandoo grau de organização, isto é, quanto maior a desorganização maior a entropia. Emtermos de estatística, a entropia pode ser definida como a medida de aleatoriedade dadistribuição, definida pela Equação 3.9.
H(X) = −Σni=1p(xi)log2(p(xi)) (3.9)

Capítulo 3. Técnicas de processamento de imagens 40
Em que n representa a quantidade de símbolos (e.g., valores de pixel) existentes,p(xi) a probabilidade do simbolo i ocorrer e H(X) a entropia associada à fonte desímbolos X . A entropia é medida em bits/símbolo e uma imagem pode ser vista comouma fonte de símbolos. O termo símbolo, nesse contexto, é usado para representaro valor do pixel. Como resultado de um processo aleatório, mede-se a probabilidadede encontrar um pixel de uma determinada cor na imagem. Dessa forma, se umaimagem possui apenas uma cor a entropia dela será -1.0 * log2(1.0) = 0. Se ela tiverduas cores a entropia é -0.5 log2(1/2) - 0.5 log2(1/2) = -log2(1/2) = 1. Quando todas asintensidades (cores) têm a mesma probabilidade de acontecer a imagem tem o valormáximo de entropia. Baseado nisso, os algoritmos de binarização entrópicos foramdesenvolvidos (PARKER, 2011).

41
4 Binarização de imagens de cromossomos
A função dos algoritmos de binarização é, basicamente, encontrar o limiar ideal,ou seja, o valor do limiar que separa corretamente os cromossomos do fundo. Nestecapítulo serão descritos os 15 algoritmos de binarização comumente utilizados (masnão na segmentação de cromossomos), e que foram avaliados neste trabalho. A Figura19 mostra os algoritmos divididos conforme a categoria a qual pertencem, segundo osurvey de Sezgin e Sankur (2004). Os algoritmos Seleção iterativa, Mean gray levele Percentage of black não foram categorizados no survey mas foram classificadosnesse trabalho como algoritmo de agrupamento e algoritmos baseados na forma dohistograma, respectivamente.
Figura 19 – Algoritmos divididos em categorias
Elaborada pela autora

Capítulo 4. Binarização de imagens de cromossomos 42
4.1 Algoritmos de agrupamento (clustering)
4.1.1 Otsu
O algoritmo de Otsu é classificado como método de agrupamento (clusters)e é um dos métodos de limiarização mais referenciados, segundo Sezgin e Sankur(2004). Esse método é caracterizado por ser não parametrizado e não supervisionado.O limiar ótimo é escolhido por critério discriminante, visando maximizar a separaçãoentre as classes resultantes (OTSU, 1979). Para isso é sugerido minimizar a somaponderada das variações dentro da classes, no primeiro plano e no fundo da imagem,obtendo o limiar. Nesse método a minimização das variações é equivalente à maxi-mização da dispersão/variância entre as classes (SEZGIN; SANKUR, 2004). O limiarótimo pode ser encontrado atráves da maximização de uma das seguintes funçõescritérios representadas pelas equações 4.1 a 4.3, onde σ2
w é a variância dentro daclasse, σ2
b corresponde à variância entre classes e σ2t é a variância total.
λ =σ2b
σ2w
(4.1)
η =σ2b
σ2t
(4.2)
k =σ2t
σ2w
(4.3)
Das funções critérios citadas η é a mais simples, logo o limiar ideal pode serobtido calculando o valor de k que maximize η ou semelhantemente maximiza σ2
b con-forme as equações.
η(k)
=σ2b
(k)
σ2t
, (4.4)
σ2b (k) =
[µtω(k)− µ(k)]2
ω(k)[1− ω(k)], (4.5)
onde µt é a média de níveis de cinza total da imagem e ω(k) e µ(k) são, respectiva-mente, os momentos cumulativos zero e de primeira ordem do histograma até o nível k.O limiar ótimo (k*) é dado como:
σ2b (k
∗) = maxσ2b (k) (4.6)

Capítulo 4. Binarização de imagens de cromossomos 43
onde 1 ≤ k < L, em que L representa a quantidade de níveis de cinza (OTSU,1979). O método de Otsu pode ser utilizado para outros objetivos além de calcular olimiar ótimo, como estimar níveis médios das classes e determinar a separabilidadedas classes. Porém, ele não é indicado para imagens que possuem variâncias do nívelde cinza do primeiro plano e fundo da imagem muito diferentes.
4.1.2 Riddler
O Algoritmo de Riddler é baseado em um esquema iterativo que mistura modelosde duas classes gaussianas. Um novo limiar Tn é escolhido a cada iteração baseadona média do primeiro plano e do fundo da imagem. As iterações são interrompidasquando o módulo da diferença entre o valor do limiar de duas iterações seguidas( | Tn − Tn+1| ) se torna suficientemente pequeno, definindo dessa forma o valor dolimiar (RIDLER; CALVARD, 1978).
4.1.3 K-means
O algoritmo K-means é um algoritmo simples de aprendizagem não supervi-sionado. A ideia principal é definir um número k de centróides, um para cada grupo.Em seguida, cada dado (nesse caso pixel da imagem) é analisado e associado aocentróide mais próximo a ele até que não tenha nenhum dado pendente formando osprimeiros grupos. Em seguida os k centróides são recalculados e os pixels devem sernovamente associados com os k centróides mais próximo a ele. Esses passos sãorepetidos até que os centróides não se movimentem mais. A localização inicial doscentróides é de grande importância pois tem influência nos resultados. A quantidadede iterações do algoritmo pode minimizar o impacto de uma inicialização errada. Oalgoritmo tem como objetivo minimizar a função objetivo, nesse caso a função de erroquadrático dada pela Equação 4.7
F =k∑j=1
x∑i=1
| x(j)i − wj |2, (4.7)
onde | xi − wj |2 é a medida de distância escolhida entre um ponto do dado xi e ocentróide wj do grupo em que ele está associado. É um indicador dos n pontos dosdados de seus respectivos centros do grupos (MACQUEEN, 1967).
4.1.4 Seleção iterativa
O método de Seleção iterativa utiliza um processo iterativo para encontrar olimiar ótimo. O método assume, arbitrariamente, um valor inicial para o limiar e refinaesse limiar de acordo com os níveis de cada classe. Inicialmente o valor de cinza médioé atribuído ao limiar e com base nesse limiar são coletadas estatísticas dos níveis

Capítulo 4. Binarização de imagens de cromossomos 44
binários (preto e branco) das classes. A partir disso, calcula-se a média da escalade cinza para os pontos abaixo do limiar Tb da mesma forma para os pontos acimado limiar (To). Utilizando esses dois parâmetros, o novo limiar é calculado conformeEquação 4.8
Tk =Tb + To
2(4.8)
Esse processo é repetido até que não haja alterações no limiar TK entre umpasso e outro (PARKER, 2011). Quando implementado em hardware o método deseleção iterativa apresenta um bom desempenho. Quando implementado em softwareé necessário utilizar o histograma da imagem para melhorar o desempenho, issoé possível devido a estrutura unidimensional do histograma. A implementação porsoftware utiliza a seguinte equação:
Tk =ΣTk−1
i=0 i.h[i]
2ΣTk−1
i=0 h[i]+
Σ255j=Tk−1+1j.h[j]
2Σ255j=Tk−1+1h[j]
(4.9)
onde h é o histograma em níveis de cinza da imagem e quando Tk = tk + 1, então tké o limiar adequado.
4.2 Algoritmos de binarização baseados em entropia
4.2.1 Kapur
O algoritmo de Kapur, categorizado como de limiar entrópico, considera quea imagem contém duas fontes de sinais diferentes e que ela é resultado de duasdistribuições de probabilidade. Então, para obter o limiar ótimo é calculada a entropiatotal de cada imagem particionada (primeiro plano e fundo) e em seguida é feita amaximização da entropia entre classes (POLETTI et al., 2012) conforme Equações4.10 e 4.11.
H(0, t) =t−1∑i=0
p− iω0
lnpi
ω0
, ω0 = Σt−1i=0pi (4.10)
H(t, L) = −L−1∑i=t
p− iω1
lnpi
ω1
, ω1 = ΣL−1i=t pi (4.11)
A entropia entre classes (Ebc) é definida conforme equação 4.12:

Capítulo 4. Binarização de imagens de cromossomos 45
Ebc(t) = H(0, t) +H(t, L) (4.12)
Sendo o limiar ótimo (topt) o valor de t que maximiza a entropia entre as classes,expresso matematicamente pela Equação 4.13.
topt = argmax{Ebc(t)
}(4.13)
4.2.2 Pun
O algoritmo de Pun considera o histograma de níveis de cinza de uma imagemcomo uma fonte de i símbolos (no caso de imagens, tons de cores), onde todosos símbolos são estatisticamente independentes. Dado que t é o valor do limiar,as duas entropias a posteriori abaixo são definidas para os pixels pretos (HB) ebrancos(Hw), onde pi é a probabilidade do nível de cinza i ocorrer (SAHOO et al.,1981).
HB = −Σti=0pilog2pi (4.14)
HW = −Σ255i=t+1pilog2pi (4.15)
Para uma imagem com níveis de cinza de 0 a 255 ele tenta achar o limiar t quemaximiza H = HB +HW .
4.2.3 Johannsen
No algoritmo de Johannsen, o objetivo é dividir a imagem minimizando a in-terdependência entre os níveis de cinza. Para isso, semelhantemente aos algoritmosde Kapur e Pun, são calculadas a entropia dos pixels pretos (HB) e separadamente,a entropia dos pixels brancos (HW). Para uma imagem com 256 níveis de cinza, aentropia associada aos pixels pretos de imagem limiarizada por um limiar t é dada pelaEquação 4.16.
HB(t) = log(Σti=0pi) +
1
Σti=0pi
[E(pt) + E(Σt−1
i=0pi)]
(4.16)

Capítulo 4. Binarização de imagens de cromossomos 46
Analogamente a entropia dos pixels brancos é dada pela Equação 4.17
Hw(t) = log(Σ255i=tpi) +
1
Σ255i=tpi
[E(pt) + E(Σ255
i=t+1pi)], (4.17)
onde E(x) = −xlog(x) e t é o valor do limiar. Note que os valores encontradospara Hb(t) e Hw(t) devem ser desprezados quando Pt = 0. Segundo o método deJohannsen, o limiar ótimo é o valor de t que minimiza a somaHB(t)+HW (t) (FACON,2016).
4.2.4 Sahoo
O algoritmo Sahoo utiliza a entropia de Reny para encontrar o limiar ótimo. Essemétodo usa duas distribuições de probabilidade (objeto e fundo) derivados da imagemoriginal em níveis de cinza, incluindo o método da soma das máximas probabilidades(Kapur) e o método entrópico de correlação. Considere p0, p1, p2, ....p255 como a dis-tribuição de probabilidade para cada nível de cinza. Desta distribuição de probabilidade,duas distribuições são derivadas, uma delas é a distribuição da classe do objeto Λ1 e aoutra da classe do fundo Λ2 dadas por:
Λ1 :p0
p(Λ1),p1
p(Λ1), ...
ptp(Λ1)
(4.18)
e
Λ2 :pt+1
p(Λ2),pt+2
p(Λ2), ...
p255
p(Λ2), (4.19)
onde p(Λ1) = Σti=0pi, p(Λ2) = Σ255
i=t+1pi e p(Λ1) + p(Λ2) = 1.
A priori a entropia de Reny de ordem α de uma imagem é definida como
HαT =
1
1− αln
255∑k=0
p(k)α, (4.20)
onde α(6= 1) é um número real positivo. A entropia de Reny associada com a distribui-ção do objeto e do fundo é dada pelas Equações 4.21 e 4.22, respectivamente.
HαΛ1(t) =
1
1− αln
t∑i=0
( pip(Λ1)
)α (4.21)

Capítulo 4. Binarização de imagens de cromossomos 47
HαΛ2(t) =
1
1− αln
255∑i=t+1
( pip(Λ2)
)α (4.22)
Seja t∗(α) o nível de cinza que maximiza HαΛ1(t) +Hα
Λ2(t), isto é,
t∗(α) = Argmax{Hα
Λ1(t) +HαΛ2(t)
}(4.23)
.
t∗(α) está em função de α. Foi observado em simulações computacionais que
t∗(α) =
t∗1 se
0 < α < 1
t∗2 se
α −→ 1
t∗3 se
1 < α <∞
(4.24)
onde t∗1, t∗2 e t∗3 são valores de cinza distintos. Quando α aproxima-se de 1 o valorde cinza t∗2 é o mesmo valor do limiar ótimo encontrado usando a soma da máximaentropia e quando α > 1 o valor de cinza de t∗3 é o mesmo limiar ótimo obtido pelométodo de entrópico de correlação. O valor do limiar ótimo deste método t∗c é calculadoutilizando os três valores de cinza t∗1, t∗2 e t∗3 pela seguinte fórmula:
t∗c = t[1]
[ap(t[1]) + 1
4ωβ1
]+
1
4t[2]ωβ2 + t3
[1− p(t[3]) + 1
4ωβ3
], (4.25)
onde t[1], t[2] t[3] correspondem às estatísticas de ordem dos valores de cinza e
t∗1, t∗2 e t∗3, p(t) =∑t
i=1 pi, ω = p(t[t3])− p(t[1]) e

Capítulo 4. Binarização de imagens de cromossomos 48
(β1, β2, β3) =
(1, 2, 1) se
| t[1] − t[2] |≤ 5 e
| t[2] − t[3] |≤ 5,
(1, 2, 1) se
| t[1] − t[2] |> 5 e
| t[2] − t[3] |> 5,
(0, 1, 3) se
| t[1] − t[2] |≤ 5 e
| t[2] − t[3] |> 5,
(3, 1, 0) se
| t[1] − t[2] |> 5 e
| t[2] − t[3] |≤ 5.
(4.26)
O valor do limiar ideal t∗c de uma imagem depende da média ponderadade t∗1, t∗2 e t∗3, e assim
min{t∗1, t
∗2, t
∗3
}≤ t∗c ≤ max
{t∗1, t
∗2, t
∗3
}, (4.27)
isso é, t[1] ≤ t∗c ≤ t[3]. Isto indica que, mesmo que o método de soma de entropiamáxima e o método de correlação de entropia não forneçam um bom valor de limiarpara uma imagem, o algoritmo de Sahho pode fornecê-lo (SAHOO; WILKINS; YEAGER,1997).
4.3 Algoritmos baseados na forma do histograma
4.3.1 Two peaks
A utilização do histograma para determinar o limiar é uma técnica bem co-mum. Foi observado que quando há um vale entre dois picos no histograma, o limiaré facilmente encontrado. O método que utiliza essa característica do histograma édenominado Two peaks. Esse método pode ser dividido basicamente em dois pas-sos: encontrar os dois picos e encontrar o menor ponto entre eles. Para encontrar oprimeiro pico é preciso descobrir o nível da escala de cinza que possui maior valor.Porém, a busca pelo segundo maior pico, provavelmente, será o pico a direita mais pró-ximo do maior ao invés de ser o segundo maior pico, o que dificulta um pouco a buscapelo segundo pico. Uma técnica simples para encontrar o segundo pico é multiplicar osvalores do histograma pelo quadrado da distância do primeiro pico (PARKER, 2011).

Capítulo 4. Binarização de imagens de cromossomos 49
Dessa forma, dado que o maior pico é o do nível j, a seleção do segundo pico é dadaconforme a Equação 4.28.
max{
(k − j)2h[k]|0 ≤ k ≤ 255}, (4.28)
onde h representa o histograma e existem 256 níveis de cinza (0-255).
4.3.2 Mean gray level
Um dos algoritmos mais simples e de fácil execução utilizado para cálculo dolimiar é o algoritmo Mean gray level (ou, nível de cinza médio). Ele é aplicado somando-se todos os valores de todos os pixels da imagem, calculando a média deles obtendoassim o limiar. Em outras palavras, o limiar é definido pelo valor médio do histogramaem tons de cinza (ROCHA, 2007). Uma variação para esse método usa a media−C ,onde C é uma constante. De maneira que, se o valor do pixel for maior que a médiasubtraída de C ele é considerado como pertencente ao objeto, senão é consideradofundo (background). A aplicação do Mean gray level faz com que, aproximadamente,metade dos pixels da imagem se tornem pretos e metade se tornem brancos.
4.3.3 Percentage of black
A fixação da porcentagem de pixels pretos em 50% pode tornar o algoritmoMean gray level falho, pois poucas imagens são 50% pretas. Porém, a ideia de fixara porcentagem de pixels pretos pode ser útil em alguns casos, como por exemploimagens de texto. A porcentagem de pixels pretos em imagens de texto variam de8,46% a 15,67%, portanto um algoritmo de limiarização que utilizasse uma taxa de 15%de pixels pretos, para essse tipo de imagem, pode obter um sucesso razoável. Baseadonisso o algoritmo Percetage of black (ou, porcentagem de preto) calcula o limiar, comauxílio de um histograma que, nesse contexto, é um vetor com mesmo número dedimensões de uma imagem em níveis de cinza. Multiplicando-se um histograma pelaquantidade de pixels total da imagem pode-se obter o número total de pixels pretos.Para encontrar o limiar conta-se o número de pixels consecutivamente a começar dopixel 0 do histograma até atingir o número desejado de pixels pretos. O limiar é o nívelde cinza associado ao último pixel contado (PARKER, 2011).
4.4 Características locais
4.4.1 Bernsen
O método de Bernsen utiliza o valor do contraste local para definir os limiares.Nele são calculadas a média entre o valor mínimo (Imin) e o máximo (Imax) de níveisde cinza que pertencem à vizinhança do pixel, conforme Equação 4.29.

Capítulo 4. Binarização de imagens de cromossomos 50
T (x, y) =Imax + Imin
2(4.29)
Porém, essa atribuição é baseada no valor de contraste local sendo expressacomo
T (x, y) =
Imax+Imin
2Imax − Imin > T
GT Imax − Imin < T(4.30)
onde T é o limiar de contraste e GT é o valor do limiar global calculado com o métodode Otsu na imagem inteira (SINGH et al., 2012).
4.4.2 Niblack
A ideia do algoritmo de Niblack é encontrar o limiar com relação ao contrasteda vizinhança. Para isso é feita uma varredura na imagem com uma janela de tama-nho N X N e para cada pixel da imagem é calculada a média µ(x, y) e o desviopadrão σ(x, y) da vizinhança contida dentro da janela. A Equação 4.31 expressa ocálculo para obtenção do limiar de cada ponto T (p) da imagem, onde µ(p) e σ(p) re-presentam a média e o desvio padrão, respectivamente, do ponto p dos pixels encon-trados na máscara e C é uma constante que representa o quanto o desvio padrão vaiinfluenciar no limiar.
T (p) = µ(p) + C ∗ σ(p) (4.31)
Na aplicação deste trabalho, se o valor do pixel é menor que T (p) ele é consi-derado cromossomo caso não, fundo. Esse método prioriza os contrastes das regiõesda imagem e é ideal para imagens que sofreram influência de sombras e iluminação.Porém quando a imagem tem uma grande variação de níveis de cinza, o uso de uma ja-nela pequena pode gerar a inclusão de ruídos na imagem. Além disso ele é paramétricoe bastante sensível ao peso (IKEDA, 2011).
4.4.3 Sauvola
O algoritmo de Sauvola é um melhoramento do método de Niblack, semelhan-temente a este, ele utiliza a média e o desvio padrão da vizinhança do pixel ao qualestá sendo calculado o limiar. Mas o algoritmo de Sauvola difere na contribuição dodesvio padrão ao resultado sendo ideal quando há uma má distribuição da iluminaçãona imagem. Quando há muito ruído na imagem, o limiar é reduzido (SEZGIN; SANKUR,

Capítulo 4. Binarização de imagens de cromossomos 51
2004). Ele adiciona um novo parâmetro ao cálculo do limiar chamado faixa dinâmicado desvio padrão. A inclusão desse parâmetro diminui a sensibilidade relacionada aopeso do método Niblack, porém torna o algoritmo sensível ao novo parâmetro (IKEDA,2011). Matematicamente, expresso pela Equação 4.32.
T (p) = µ(p) ∗ [1 + C ∗ (σ(p)
R− 1)] (4.32)
A média e o desvio padrão são calculados utilizando os pixels vizinhos con-tidos na janela e C e R os parâmetros de peso e faixa dinâmica do desvio padrão,respectivamente.
4.4.4 White
O algoritmo White foi desenvolvido para aplicações de binarização de documentovisando facilitar o Optical Character Recognition (OCR). Este é um pouco mais simplesque os citados acima, pois realiza uma simples comparação do valor do tom de cinzado pixel com a média dos tons de cinza dos seus vizinhos. Na aplicação deste trabalho,se o pixel é significativamente mais claro que a média, ele pertence à classe doscromossomos, senão pertence ao fundo. O método White é bastante sensível ao seuparâmetro bias, que pode trazer ótimos resultados a depender do bias escolhido paracada imagem.
T (p) =µ(p)
bias(4.33)

52
5 Materiais e metodologia dos experimentos
Neste capítulo serão descritas as ferramentas utilizadas na realização destetrabalho, assim como, as etapas utilizadas para concretização dos objetivos.
Para alcance dos objetivos foi utilizada a linguagem c++ no software MicrosoftVisual Studio Community 2015 que possui licença gratuita para desenvolvedoresindividuais e pesquisa acadêmica. Junto a ele, foi utilizada a biblioteca OpenCV (OpenSource Computer Vision Library ) construída para fornecer uma infra-estrutura comumpara aplicações com visão computacional e o software MatLab na versão R2013busado para testar os algoritmos de binarização. Além do Photoshop Portable PSCS6utilizado na geração das imagens rotuladas de ambas as bases, com auxílio do CRCN-NE na base por eles fornecida. Os experimentos foram realizados em um notebookDell vostro 5470 Intel(R) core(TM) i7-4510U CPU @ 2.00GHz com memória RAM de8GB, disco rígido de 500GB, placa de vídeo NVIDIA GEFORCE dedicada de 2GB ecom sistema operacional Windows 8.1.
A metodologia utilizada pode ser dividida basicamente em cinco etapas: i) verifi-cação de resultados, utilização de métricas avaliativas de segmentação; ii) avaliaçãoparamétrica, em que os algoritmos de binarização parametrizados serão testados afim de encontrar os parâmetros mais adequados para cada base; iii) segmentação,as imagens serão binarizadas a fim de evidenciar os objetos de interesse; iv) pósprocessamento, para melhorar a qualidade da segmentação; v) contagem, verificaçãode quantos objetos (componentes conexos) há em cada imagem. As etapas serãomelhores descritas nas próximas subseções.
5.1 Verificação dos resultados
Para comparar os algoritmos e então descobrir/classificar qual o mais adequadopara segmentar cromossomos, ambas as bases foram segmentadas manualmentecom auxílio dos funcionários/especialistas do CRCN, utilizando uma ferramenta dophotoshop portable, chamada varinha mágica. Essa ferramenta permite a fácil seleçãode objetos na imagem, facilitando a binarização das imagens manualmente. Dessaforma, cada imagem da base terá uma correspondente com segmentação ideal, comomostra a Figura 20. Então, as imagens geradas pelos algoritmos podem ser comparadase avaliadas conforme as métricas descritas abaixo.

Capítulo 5. Materiais e metodologia dos experimentos 53
Figura 20 – Exemplo base CRCN (a) imagem original (b) segmentação ideal (ou imagemrotulada)
Elaborada pela autora
5.1.1 Métricas avaliativas
A avaliação da segmentação é realizada comparando pixel a pixel, onde cadapixel da imagem gerada pelo algoritmo é comparado ao pixel de localização corres-pondente da imagem rotulada (ideal). Como as imagens estão binarizadas os valoresque os pixels podem assumir são 0 (preto) ou 1 (branco). Os resultados das compa-rações são armazenados em uma matriz chamada matriz de confusão. A matriz deconfusão, também conhecida como matriz de erro, é uma das técnicas mais utilizadaspara avaliação de classificação de dados pois a partir dela é possível calcular váriasmétricas descritivas e analíticas. Trata-se de uma matriz quadrada, no caso de imagensbinárias possui dimensão 2x2, em que geralmente as colunas representam os dadosde referência (imagem modelo/rotulada) e as linhas, os dados gerados pelo algoritmo aser avaliado. A Figura 21 mostra um exemplo do uso da matriz de confusão (considerecada quadrado como sendo um pixel). A Figura 21a exemplifica uma segmentaçãoideal, a Figura 21b é um exemplo de uma imagem segmentada gerada pelo algoritmoavaliado e a Figura 21c mostra a matriz de confusão correspondente à comparaçãoentre elas.

Capítulo 5. Materiais e metodologia dos experimentos 54
Figura 21 – a) Segmentação ideal (b) Imagem correspondente resultante de algoritmo avaliado(c) Matriz de confusão
Elaborada pela autora
Os valores armazenados na diagonal principal, com as coordenadas (1,1) e(2,2) correspondem respectivamente aos verdadeiros positivos (True Positive - TP)e verdadeiros negativos (True Negative - TN) e referem-se à quantidade de pixelsclassificados corretamente. E os valores armazenados na diagonal secundária, comas coordenadas (1,2) e (2,1), correspondem respectivamente aos falsos positivos(False Positive - FP) e Falsos negativos (False Negative - FN), e referem-se aos pixelsclassificados erroneamente pelo algoritmo avaliado.
Algumas das técnicas descritivas que utilizam a matriz de confusão comobase, utilizada neste trabalho são: acurácia, sensibilidade, precisão e coeficiente decorrelação de Matthews (MCC). A acurácia é a proporção de acertos (ou taxa de acerto)do algoritmo, expressa pela quantidade de pixels classificados corretamente em relaçãoao total de pixels classificados, como mostra a equação 5.1 a seguir.
acuracia =TP + TN
TP + TN + FP + FN(5.1)
Já a sensibilidade representa a quantidade de pixels classificados corretamenteem relação aos pixels (ou região da imagem) rotulados pelo especialista (MARCOMINI;SCHIABEL; VERÇOSA, 2012). Ela define a capacidade do algoritmo de identificarcorretamente os pixels do primeiro plano da imagem. Definido matematicamente como:
sensibilidade =TP
TP + FN(5.2)
A Precisão, também chamada de predição de valores positivos (Positive Predic-tive Value - PPV), é a quantidade de pixels classificados como sendo cromossomos,sobre o número total de pixels que o algoritmo identificou como cromossomos, conformeequação 5.3. Esta métrica é mais útil que a especificidade quando a quantidade de

Capítulo 5. Materiais e metodologia dos experimentos 55
pixels pertencentes às duas classes (preto e branco) são muito diferentes pois, quandohá um desbalanceamento entre as classes, a especificidade para todos os métodostende a ser próxima de 1, não fazendo sentido (POLETTI et al., 2012).
precisao = PPV =TP
TP + FP(5.3)
O coeficiente de correlação de Matthews (MCC) é considerado uma boa métricapara avaliar a similaridade entre classificações binárias (POLETTI et al., 2012) e podeser usado com aplicações não binárias também. Diferente das métricas citadas acima,compreende um intervalo entre -1 e +1. O valor -1 indica total desacordo entre asimagens, e o valor +1 indica total combinação entre elas. Quando o coeficiente decorrelação é igual a 0 significa que há aleatoriedade na predição dos valores (BALDI etal., 2000). Pode ser calculado pela expressão abaixo.
MCC =TP × TN − FP × FN√
(TP + FP )(TP + FN)(TN + FP )(TN + FN)(5.4)
Existem situações em que a avaliação obtida através do coeficiente de corre-lação de Matthews não é coerente. Por exemplo, se o algoritmo predizer poucos ounenhum elemento falso positivo e ao mesmo tempo poucos elementos verdadeirospositivos o MCC será relativamente alto (BALDI et al., 2000).
As características de aplicação de cada métrica citada acima serão melhorexplicadas na Seção 6.2. As métricas foram calculadas utilizando o código Evaluate emMatlab, disponível no MathWorks (https://www.mathworks.com/matlabcentral/fileexchange/37758-performance-measures-for-classification/content/Evaluate.m). Nesse código,foi adicionado o cálculo do coeficiente de correlação de Matthews.
5.1.2 Aquisições das imagens
Para testes e validação do algoritmo desenvolvido, foram utilizadas 2 bases deimagens diferentes. A primeira base de imagem utilizada foi produzida e disponibilizadapelo Laboratório de Dosimetria Biológica do CRCN-NE e consiste de 35 imagens (videFigura 22) de células em metáfase, que foram capturadas através de um microscó-pio óptico, rotuladas por especialistas e estão apresentadas no formato TIFF, comresolução de 2048 x 1536 pixels.

Capítulo 5. Materiais e metodologia dos experimentos 56
Figura 22 – Exemplo da base de imagens produzidas e fornecidas pelo CRCN-NE
Na aquisição das imagens, foram coletados 10 ml de amostras de sangueperiférico por punção venosa de um doador saudável, não fumante e que nos últimosseis meses, antes da coleta, não tenha sido exposto à radiação terapêutica, raioX diagnóstico, vacinação viral ou tenha consumido drogas ilícitas. Essa amostra foiexposta a uma fonte 60Co (Irradiador Gammacel 220), localizada no Departamento deEnergia Nuclear (DEN-UFPE), e em seguida foram mantidas na estufa 37ºC por 2 horas,tempo de espera para que o mecanismo de defesa e reparação celular possa atuar, parao cultivo de células e preparação das lâminas. As preparações citológicas para análisecromossômica foram obtidas a partir da cultura de linfócitos onde foram adicionados 4ml de meio RPMI 1640 (Sigma - Aldrich) suplementado com 1 ml de soro bovino fetal(Biological Industries), 0,2 ml de fitohemaglutinina. Em seguida, os frascos forammantidos na estufa a 37 °C, por 48 horas. Após 46 horas, foi adicionado aos frascos0,1 ml de colchicina na concentração de 0,0016%. Ao completar 48 horas de cultivo,o material foi centrifugado por 6 minutos a 1800 rpm. Para a preparação das lâminas,foram realizadas centrifugações e trocas de fixador para que o conteúdo da culturaestivesse transparente. Após o processo de fixação, foram confeccionadas lâminasa partir do precipitado de células ressuspenso em 0,5 – 0,75 ml de solução fixadora.O precipitado de células ressuspenso foi gotejado em dois pontos em cada lâmina eessas foram colocadas para secar a temperatura ambiente durante 24 horas (MENDES,2014). Em seguida, as lâminas foram coradas com Giemsa a 5% durante 7 min eentão, as fotos das lâminas foram capturadas, atráves de uma câmera acoplada a ummicroscópio óptico - Leica DM 1000 (vide Figura 23 ).

Capítulo 5. Materiais e metodologia dos experimentos 57
Figura 23 – Microscópio óptico com câmera acoplada utilizado para aquisição das imagens
Fotografado pela autora
A segunda base de imagem consiste em 162 imagens de cromossomos, adqui-rida de 117 células obtidas durante a prometáfase, com seus respectivos cariogramas(vide exemplos na Figura 24). Os cariogramas foram feitos manualmente por um espe-cialista em citologia. As imagens foram capturadas por meio de um microscópio ópticoapós terem sido corados com quinacrina (Bandeamento Q). Elas apresentam formatoBPM, com tamanho de 768x576 e resolução de 8 bits/pixels. Essas imagens foram dis-ponibilizadas pelo Laboratório de Imagiologia Biomédica (BioImLab) do Departamentode Engenharia da Informação da universidade de Padova, localizado na Itália (http://bioimlab.dei.unipd.it/Chromosome%20Data%20Set%204Seg.htm).
Figura 24 – Exemplo da base de imagem. Cromossomos em prometáfase (a) e seu respectivocariograma (b)
BioImLab (http://bioimlab.dei.unipd.it/Chromosome%20Data%20Set%204Seg.htm)
Algumas células possuem mais de uma imagem que foram capturadas de dife-rentes ângulos. Essas não foram utilizadas nesse trabalho, sendo assim foram utilizadas77 imagens, das 162 disponibilizadas pelo BioImLab.

Capítulo 5. Materiais e metodologia dos experimentos 58
5.2 Avaliação paramétrica
Dos 15 algoritmos testados, quatro deles possuem parâmetros (Bernsen, Ni-black, Sauvola e K-means). Sendo assim, para obter os melhores resultados de cadaalgoritmo, para cada base de dados, eles foram executados várias vezes com os pa-râmetros sendo variados em intervalos próximos aos parâmetros default (padrão) decada algoritmo.
O algoritmo de Bernsen classificado como local, possui dois parâmetros: otamanho da janela e quantidade de contraste. O valor padrão para esse algoritmo é 31para janela e 15 para o contraste. Os valores de janela testados foram 9, 15 e 31 e naquantidade de contraste foram 5, 10, 15 e 20. Dessa forma, o algoritmo foi executado12 vezes com a combinação desses parâmetros, isto quer dizer que ele foi executadouma vez com tamanho de janela igual a 9 e contraste igual a 5, em seguida, novamentecom tamanho de janela igual a 9 mas, com contraste igual 10 e assim sucessivamenteexecutando todas as possibilidades.
O algoritmo de Niblack, semelhantemente ao de Bernsen, também classificadocomo local possui dois parâmetros. São eles: o tamanho da janela e o peso, sendoseus valores padrão 31 e -0.8, respectivamente. Os valores de janela avaliados foram9, 15 e 31 e de peso foram -0.8, -0.2, 0.2 e 0.8 combinados 2 a 2. O algoritmo foiexecutado 12 vezes.
O algoritmo Sauvola, também local, tem apenas o tamanho da janela comoparâmetro. O tamanho da janela foi variado da mesma forma que os citados acima 9, 15e 31, sendo executados apenas 3 vezes. O algoritmo de K-means, o único algoritmo deagrupamento, tem como parâmetro o número de grupos denominado como k. Tambémfoi executado 3 vezes com os valores de k igual 3, 5 e 7 em cada execução. O númeromáximo de iterações utilizado foi de 10 iterações. Esta quantidade foi suficiente paraobter bons resultados.
As imagens resultantes obtidas, foram avaliadas conforme Subseção 5.1 e osparâmetros que obtiveram melhores resultados foram comparados com os demaisalgoritmos não parametrizados. É importante ressaltar que essa etapa foi realizadaduas vezes, uma para cada base de imagens, encontrando diferentes melhores valorespara cada algoritmo. Os melhores parâmetros para cada algoritmo e cada base deimagem serão mostrados na Seção 6.1.
5.3 Segmentação
Na etapa de segmentação foi utilizada a técnica de binarização descrita naSubseção 3.3.1. A maior dificuldade na aplicação da binarização é a definição do valordo limiar que separa corretamente as duas classes de pixels (preto e branco). Por isso,

Capítulo 5. Materiais e metodologia dos experimentos 59
neste trabalho foram executados e avaliados 15 algoritmos de binarização (descritosno Capítulo 4), implementados em Matlab, com diferentes estratégias para encontrar olimiar ideal.
Eles foram divididos em categorias quanto a forma de explorar os dados daimagem, são eles: Otsu, K-means, Riddler e Seleção iterativa, classificados comoalgoritmos de agrupamento; Kapur, Pun, Johannsen e Sahoo, classificados como algo-ritmos baseados em informações de entropia; Two peaks, Mean gray level e percentageof black, categorizados como algoritmos baseados na forma do histograma; Bernsen,Niblack, Sauvola e White classificados como algoritmos baseado em característicaslocais. Esses Algoritmos são bem conhecidos mas, com exceção do algoritmo de Otsu eK-means, eles não são muito aplicados no contexto de segmentação de cromossomos.
Os algoritmos foram avaliados utilizando quatro métricas: coeficiente de correla-ção de Matthews (MCC), precisão, sensibilidade e acurácia. Aqueles que obtiverammaior valor de MCC e precisão, nessa ordem, e resultados razoáveis para sensibilidadee acurácia foram considerados os melhores. Todos eles foram executados em duasbases de imagens com diferentes características, a fim de descobrir quais os melhoresalgoritmos para cada base de imagens. Ao fim dessa etapa, os melhores algoritmospara cada base, foram classificados/encontrados e suas imagens resultantes seguirampara a próxima etapa. Exemplos de imagens resultantes de cada algoritmo em cadabase de imagem pode ser visto nos Apêndices A e B.
5.4 Pós processamento
As imagens advindas do algoritmo de binarização não ficaram muito próximasà segmentação ideal feita manualmente, interferindo na qualidade das etapas declassificação posteriores. Por isso, foi necessário a utilização de técnicas de pósprocessamento a fim de melhorar a qualidade da segmentação eliminando os pequenosruídos que permaneceram ou foram geradas na binarização.
As técnicas utilizadas para redução de ruídos aplicados nas imagens resultantesdos melhores algoritmos foram o filtro gaussiano e a operação morfológica de abertura,seus parâmetros também foram variados para encontrar qual o mais adequado. Ofiltro gaussiano foi usado para suavizar a imagem e apagar os pequenos detalhes daimagem. A operação de abertura foi aplicado após a aplicação do filtro gaussiano. Ele foiutilizado para remover os pequenos ruídos que surgiram na segmentação da imagem,sem que as propriedades de forma do cromossomos fossem alteradas. Os parâmetrostestados no pós processamento foram o tamanho da janela do filtro gaussiano e atamanho do elemento estruturante da abertura. Para janela foram testados os valores3, 9 e 15 e para o tamanho do elemento estruturante foram testados os valores 5, 7 e 9.O algoritmo foi executado 9 vezes com a combinação desses parâmetros dois a dois.

Capítulo 5. Materiais e metodologia dos experimentos 60
O elemento estruturante utilizado tem a forma elíptica, por ser mais próximo ao formatode um cromossomo. O pós processamento foi feito em c++ com auxílio da bibliotecaOpencv. Vide código 5.1 abaixo.
Código 5.1 – Código reponsável pelo pós processamento
Mat src , out , ds t ;GaussianBlur ( src , out , Size (3 , 3) , 0 , 0 , BORDER_DEFAULT) ;morph_size = 5;Mat element = getS t ruc tu r ingE lement (MORPH_ELLIPSE, Size (2 *
morph_size + 1 , 2 * morph_size + 1) ) ;morphologyEx ( out , dst , MORPH_OPEN, element ) ;
No código acima, a variável src (Mat) contém a imagem resultante da segmenta-ção que será processada e a variável out (Mat) a imagem resultante do filtro gaussiano.O terceiro parâmetro refere-se ao tamanho da janela, nesse caso 3x3. O quarto e quintoparâmetros referem-se ao desvio padrão na direção x e y, respectivamente. Quandoambos são zero eles são computados pelo tamanho da janela. Na aplicação da opera-ção morfológica de abertura é utilizada a função morphologyEx, que além das matrizescontendo a imagem filtrada pelo filtro gaussiano e imagem resultante, possui parâmetrosindicando qual o tipo de operação será executada e o elemento estruturante. As opçõesde operações executadas pela função morphologyEx são: abertura (MORPH_OPEN),fechamento (MORPH_CLOSE ), gradiente morfológico (MORPH_GRADIENT), Top Hat(MORPH_TOPHAT) e Black Hat (MORPH_BLACKHAT). Na função getStructuringEle-ment as possíveis formas de elemento estruturante são: retangular (MORPH_RECT),elíptico (MORPH_ELLIPSE) e cruz (MORPH_CROSS). E a variável morph_size contémo tamanho do elemento estruturante.
5.5 Contagem
Com as imagens já segmentadas, o método findContours do opencv foi utilizadopara encontrar os pontos de contornos dos componentes conexos de cada imagem.Alguns ruídos podem se parecer com objetos, e por serem grandes, não foram elimi-nados na etapa de pós processamento, dessa forma foi necessário calcular a área decada contorno (encontrado por findContours) da imagem e então classificá-los comocromossomo, ou não, de acordo com seu tamanho. O método de contagem implemen-tado possui 3 parâmetros: a imagem segmentada (Mat), um número limite inferior (int)e um número limite superior. No fim da execução do algoritmo de contagem um arquivono formato .txt é gerado contendo a quantidade de componentes conexos contidosem cada imagem. É importante ressaltar que apenas os componentes conexos quepossuem valor de área entre os limites inferior e superior passados como parâmetrossão considerados na contagem. Além do arquivo, novas imagens são geradas contendoapenas os componentes conexos que foram computados na contagem. Essa etapa foi

Capítulo 5. Materiais e metodologia dos experimentos 61
executada várias vezes com variações dos parâmetros: limite inferior e limite superior.Os valores testados na base do CRCN foram: 150 a 6000, 200 a 3000, 200 a 6000, 200a 6500, 200 a 7000, 250 a 3000, 250 a 4000, 250 a 6000, 250 a 7000 e 1000 a 6000.Os valores testados na base BioImLab foram: 0 a 8000, 50 a 9000, 100 a 8000, 200a 6000, 250 a 7000, 250 a 8000, 250 a 9000, 1000 a 6000. Para avaliar os melhoresparâmetros foi utilizado um critério expresso pela função abaixo.
f(x) = ΣNi=0(Nideali −Nresultantei) (5.5)
Na expressão acima, f(x) representa a soma das diferenças de cada imagem,ou o quanto o algoritmo contou errado, sendo N a quantidade de imagens da base,Nideali o número de componentes conexos que existem na imagem rotulada denúmero i eNresultantei o número de componentes conexos que contém na imagemresultante segmentada pelo algoritmo. Dessa forma, os parâmetros que alcançaramo menor valor f(x) foram considerados os melhores. Optou-se pela não utilizaçãodo módulo na função para que os resultados com maiores valores de Nresultanteifossem valorizados, ou seja, os erros Nresultantei > Nideali foram consideradosmelhores porque podem ser tratados em futuras etapas porém se um cromossomo forapagado Nresultantei < Nideali o erro será definitivo/não recuperável.
O método utilizado na contagem será descrito abaixo.
Código 5.2 – Código responsável pela contagem de cromossomos e filtragem de acordo com ovalor da área (c++/opencv)
vo id contagemComponente ( Mat b inary , i n t l i m I n f , i n t limMax ) {vector < i n t > i nd i ces ;vector < vector <Point >> contours ;f indContours ( b ina ry . c lone ( ) , contours , CV_RETR_EXTERNAL
, CV_CHAIN_APPROX_SIMPLE) ;
f o r ( i n t i = 0 ; i < contours . s ize ( ) ; i ++) {i n t area = contourArea ( contours [ i ] ) ;i f ( area > l i m I n f && area < limMax ) {
i nd i ces . push_back ( i ) ;areas . push_back ( area ) ;
}}Mat drawing = Mat : : zeros ( b inary . s ize ( ) , CV_8UC3) ;Scalar co l o r = Scalar (255 , 255 , 255) ;i n t numberR = 0;f o r ( i n t i = 0 ; i < i nd i ces . s ize ( ) ; i ++) {
drawContours ( drawing , contours , i nd i ces [ i ] ,co lo r , CV_FILLED , 4 , h ierarchy , 0 , Po in t ( ) ) ;
}/ / sa l va r o quantidade de contornos ind i ces . s i ze ( ) em
arqu ivo

Capítulo 5. Materiais e metodologia dos experimentos 62
/ / sa l va r imagem . .}
O método findContours do opencv é responsável por encontrar os pontos decontornos de todos os componentes conexos de uma dada imagem binária. O terceiroparâmetro desse método indica o modo de obtenção do contorno e o parâmetropassado CV_RETR_EXTERNAL indica que serão retornados apenas os contornosmais externos e extremos. Isso significa que se houver, um contorno dentro de outro, istoé, falhas e/ou buracos também conhecido como ilhas, dentro de algum cromossomo daimagem (como mostra a Figura 25) esses contornos não serão retornados pela função.Na Figura 25, por exemplo, a função retornará apenas um contorno (que é o ideal),caso seja utilizado outro parâmetro (como CV_RETR_LIST ou CV_RETR_TREE) afunção findContours retornaria três contornos. Esse parâmetro elimina o uso de técnicasadicionais para eliminar as falhas/ilhas além de permitir maior flexibilidade, ou aceitação,de valores de sensibilidade e precisão mais baixos sem prejudicar a contagem. O quartoparâmetro passado no método refere-se a forma de aproximação dos contornos e oparâmetro passado CV_CHAIN_APPROX_SIMPLE indica que apenas pontos extremosde segmentos horizontais, verticais e diagonais serão retornado no vetor de pontos.Após encontrar os componentes conexos contidos na imagem, a área (obtida pelafunção contourArea) de cada contorno é calculada e somente aqueles que estão dentrodo intervalo limInf < areaComponente < limMax são armazenados. Então,uma nova imagem binária é gerada (variável drawing no código) utilizando a funçãodrawContours, que desenha os contornos desejados, na cor especificada no parâmetrocolor (do código acima) com CV_FILLED indicando que o interior do contorno tambémserá desenhado/preenchido. Em seguida, a quantidade de contornos é inserida numarquivo e a imagem criada é salva. As imagens geradas nessa etapa também foramavaliadas com as métricas acurácia, sensibilidade, precisão e coeficiente de correlaçãode Matthews, conforme Subseção 5.5.1.
Figura 25 – Exemplo de cromossomo com buracos ou ilhas
Elaborada pela autora

63
6 Validação de experimentos (resultados)
6.1 Avaliação paramétrica
Na escolha dos parâmetros dos algoritmos parametrizados, Bernsen, Niblack,Sauvola e K-means, foram realizados alguns testes para descobrir quais valores apre-sentam melhores resultados e a influência de cada parâmetro na qualidade da seg-mentação obtida, por cada algoritmo. Em todos os algoritmos, exceto K-means, osparâmetros que obtiveram melhores resultados variaram de acordo com a base de ima-gens utilizada. Para o algoritmo de Bernsen, que possui dois parâmetros - tamanho dajanela e contraste - nota-se que, na base do CRCN, o contraste exerce maior influênciana qualidade da imagem com relação à acurácia, precisão e MCC, porém o tamanhoda janela teve maior impacto na sensibilidade, como mostra a Figura 26 abaixo.
Figura 26 – Avaliação de parâmetros no algoritmo de Bernsen CRCN
Elaborado pela autora
No gráfico da Figura 26, o eixo das abcissas representa a variação de parâme-tros, sendo o primeiro número referente ao tamanho da janela e o segundo referente aocontraste. Os parâmetros que obtiveram os melhores resultados e que serão utilizadosna próxima etapa são a combinação, tamanho de janela 9x9 e contraste 20. Embora osvalores da sensibilidade tenham sido superiores nas janelas com tamanhos maiores,as demais métricas foram inferiores.

Capítulo 6. Validação de experimentos (resultados) 64
Diferentemente da base do CRCN, na base obtida do BioImLab o tamanho dajanela teve mais influência nas métricas utilizadas do que o contraste (como mostraa Figura 27), provavelmente pelo fato de as imagens possuírem pouca variação deluminosidade e menor presença de ruídos.
Figura 27 – Avaliação de parâmetros no algoritmo de Bernsen BioImLab
Elaborado pela autora
A janela de tamanho 31x31 e contraste de 20 foram os parâmetros selecionadospara a base BioImLab. Embora tenha apresentado sensibilidade menor que os demais,eles possuem melhores resultados de acurácia, precisão e MCC.
O algoritmo de Niblack também possui dois parâmetros - o tamanho da janela eo peso. A variação de peso utilizada não influenciou muito os resultados, em ambas asbases. Na base do CRCN a variação da janela teve mais impacto na sensibilidade e noMCC, como mostra a Figura 28 abaixo.

Capítulo 6. Validação de experimentos (resultados) 65
Figura 28 – Avaliação de parâmetros no algoritmo de Niblack - CRCN
Elaborado pela autora
O tamanho da janela selecionado foi 31x31 e peso de -0.8, pois obtiveram maiorsensibilidade, precisão e MCC. Nota-se nas Figuras 28 e 29 que o aumento do tamanhoda janela está diretamente relacionado à melhora nas taxas de sensibilidade, precisãoe MCC em ambas as bases. Na base do BioImLab os parâmetros escolhidos foram dejanela 31x31 e peso de 0.8, pois também apresentaram os melhores resultados paraas quatro métricas.
Figura 29 – Avaliação de parâmetros no algoritmo de Niblack - BioImLab
Elaborado pela autora

Capítulo 6. Validação de experimentos (resultados) 66
Nota-se também, nos gráficos das Figuras 26 e 28, que os valores da precisãoe MCC foram bem baixo se comparados a acurácia e a sensibilidade provavelmentepela presença de ruídos na base de imagens que não foram removidos, pelo contrário,foram intensificados pelo algoritmo de Bernsen e Niblack. Semelhantemente, a acuráciana base do BioImLab obtidas pelos algoritmos de Bernsen e Niblack foram inferioresàs obtidas na base do CRCN pois os algoritmos retornaram imagens com o fundoinvertido em relação a segmentação ideal, o Apêndice B contêm alguns exemplosdessas imagens. Dessa forma, a precisão e o MCC, por serem as métricas que melhorcomparam a similaridade entre as imagens, também apresentaram valores bem baixos.
O algoritmo de Sauvola utilizado tem apenas o tamanho da janela como parâ-metro. Na base do CRCN há uma relação diretamente proporcional entre o tamanho dajanela e os resultados das métricas. Todas as métricas obtiveram melhores resultadosao aumentar o tamanho da janela, dessa forma, o tamanho da janela usado foi o de31x31.
Figura 30 – Avaliação de parâmetros no algoritmo de Sauvola - CRCN
Elaborado pela autora
Na base do BioImLab o algoritmo Sauvola não apresentou nenhuma tendênciaou comportamento padrão. O parâmetro selecionado foi a janela de tamanho 15x15,que obteve maior MCC e precisão, além de acurácia levemente superior. O algoritmode Sauvola, quando comparado ao demais avaliados, foi bastante afetado/influenciadopela presença de ruídos apresentando/resultando baixos valores de acurácia, precisãoe MCC na base do CRCN, e valores bem melhores na base do BioImLab, onde apresença de pequenos ruídos é praticamente inexistente.

Capítulo 6. Validação de experimentos (resultados) 67
Figura 31 – Avaliação de parâmetros no algoritmo de Sauvola - BioImLab
Elaborado pela autora
O algoritmo de K-means que tem como único parâmetro a quantidade de gruposk não obteve muita mudança com a variação desse. Tendo o valor k = 3 obtido a melhorperformance para todas as métricas, exceto para a sensibilidade a qual obteve um valorlevemente inferior. Essa comparação é mostrada no gráfico da Figura 32.
Figura 32 – Avaliação de parâmetros no algoritmo de K-means - CRCN
Elaborado pela autora

Capítulo 6. Validação de experimentos (resultados) 68
Na base BioImLab também foi utilizado o parâmetro k = 3 por possuir o maiorMCC e sensibilidade. Apesar de menor precisão e acurácia, a diferença entre osresultados foi considerada desprezível, como mostra a Figura 33.
Figura 33 – Avaliação de parâmetros no algoritmo de K-means - BioImLab
Elaborado pela autora
Após essa etapa os quatro algoritmos foram comparados com os demais nãoparametrizados utilizando os melhores parâmetros aqui descritos.
6.2 Segmentação
De forma geral, a base disponibilizada pelo CRCN obteve altos valores deacurácia (em média 87,83%), os algoritmos que obtiveram maiores valores de acu-rácia foram: White (97,87%), Johannsen (97,81%) e Seleção iterativa (97,71%). Demodo diferente, a base de dados BioImLab obteve uma média de 71,09%. Os melhoresresultados obtidos foram dos algoritmos de K-means (96,74%), Riddler (96,62%) e Twopeaks (96,08%), nessa ordem. Apesar dos bons resultados relacionados à acurácia, foiconstatado que somente a acurácia não é uma boa métrica.
Os valores de acurácia encontrados dão uma visão geral da qualidade dasegmentação gerada pelos algoritmos. Porém, é importante ressaltar que, quando háuma grande diferença entre a quantidade de dados de cada classe, ela não representabem a qualidade da segmentação.
No contexto de cromossomos, principalmente na base disponibilizada peloCRCN, os cromossomos ocupam uma pequena porção da imagem sendo o fundo

Capítulo 6. Validação de experimentos (resultados) 69
(background) muito maior. Dessa forma, imagens com alta acurácia nem semprerepresentam uma boa segmentação, como mostra a Figura 34 abaixo.
Figura 34 – Exemplo representatividade da acurácia (a) imagem rotulada (ideal) (b) imagembinarizada com K-means - 98, 97% (c) imagem binarizada com Sauvola - 47,78% (d)
imagem binarizada com White - 98, 52%
Elaborada pela autora
A Figura 34b mostra uma boa segmentação realizada pelo algoritmo de K-means e, consequentemente, uma boa acurácia (98,97%). Semelhantemente, a Figura34c mostra uma segmentação ruim com bastantes ruídos e coerentemente sua acuráciafoi bem baixa. Porém, a Figura 34d apresenta uma alta acurácia mas os cromossomos,que são o objeto de interesse desse trabalho, estão muito apagados tornando-a inviável.Por isso, foram utilizadas outras métricas que complementaram a acurácia. Ela seráutilizada de maneira a indicar quando há excesso de ruído nas imagens resultantes,classificando quais os piores algoritmos no contexto de cromossomos.
A sensibilidade avalia a predição/taxa de acerto referente ao primero plano,nesse caso os cromossomos. Ela diz o quanto um cromossomo foi classificado peloalgoritmo como cromossomo, sendo ele de fato um cromossomo (sendo classificadomanualmente como cromossomo). Porém, não leva em conta o fundo da imagem.Caso o algoritmo avaliado classifique o fundo como sendo cromossomo, esse erronão influenciará no resultado da sensibilidade, ou seja, apesar do erro obterá alta

Capítulo 6. Validação de experimentos (resultados) 70
sensibilidade. A Figura 35 abaixo exemplifica a métrica da sensibilidade.
Figura 35 – Análise Sensibilidade (a) Imagem original (b) imagem binarizada pelo algoritmo Twopeaks - 79,28% (c) imagem binarizada pelo algoritmo Johannsen - 0% (d) imagem
binarizada pelo algoritmo Pun 100%
Elaborada pela autora
A Figura 35b mostra a imagem resultante do algoritmo Two peaks com sen-sibilidade de 79,28%, sendo exemplo de boa representatividade na relação métricaqualidade de segmentação. Semelhantemente, a Figura 35c resultante do algoritmo deJohannsen obteve todos os cromossomos contidos apagados, retornando uma imagemcompletamente preta e apresentando assim, o valor minímo da métrica (zero). Porém, aFigura 35d resultante do algoritmo de Pun apresenta sensibilidade de 100%, o que nãocondiz com a qualidade da segmentação da imagem, mas como todos os cromossomosclassificados pelos especialistas como cromossomos foram classificados pelo algoritmocomo cromossomos, ele retornou alto valor de sensibilidade, não considerando os errosdo fundo da imagem. Sendo assim, também não pode ser utilizada como única métrica,mas é útil na classificação dos cromossomos. Os algoritmos com maior sensibilidadena base do CRCN são: Pun, Percentage of black e Sauvola. Já na base do BioImLabforam: Percentage of black, Otsu e Johannsen.

Capítulo 6. Validação de experimentos (resultados) 71
A precisão (o número de pixels classificados como cromossomo sobre o númerototal de pixels que o algoritmo identificou como cromossomo) possui um bom desempe-nho quando há um desequilíbrio entre as dimensões das classes em direção à classenegativa, podendo ser chamada também de taxa de descoberta de falsos (FDR). Elaobteve uma avaliação mais fiel à qualidade da segmentação que a sensibilidade. Assimcomo a sensibilidade, ela avalia o quanto o cromossomo foi classificado corretamente,porém, quanto mais o algoritmo testado classificar o fundo da imagem como sendocromossomo, menor será o valor de precisão dele. Desse modo, a sensibilidade ea precisão combinadas podem ser uma boa métrica. Os algoritmos que obtiveramos maiores valores de precisão na base do CRCN foram: Seleção iterativa, Otsu eK-means. Na base BioImLab foram: Riddler, K-means e Sahoo. No geral, a base doBioImLab obteve resultados superiores com relação à precisão.
Figura 36 – Exemplo de precisão. (a) imagem ideal (b) imagem resultante do algoritmo Seleçãoiterativa 96,82% (c) imagem resultante do algoritmo Pun 3,28% (d) imagem
resultante do algoritmo mean 98,13%
Elaborada pela autora
A Figura 36b mostra uma boa segmentação com precisão de 96,82%. A Figura

Capítulo 6. Validação de experimentos (resultados) 72
36c apresenta baixa precisão (3,28%) sendo assim segmentação bem distante do ideal.Note porém, que a Figura 36d apresenta boa precisão mas os cromossomos estão umpouco apagados o que pode interferir nas futuras etapas.
Das quatro métricas utilizadas para avaliar a qualidade da segmentação nestetrabalho, pode-se constatar que o coeficiente de correlação de Matthews é a que possuimelhor correspondência qualidade-valor, por isso, foi a principal métrica usada paradefinir o melhor algoritmo em cada base. Ela também possui, como vantagem, o fatode não ser sensível ao desequilíbrio das dimensões entre classes e é consideradauma boa métrica para classificações binárias (POLETTI et al., 2012). Os três melhoresalgoritmos testados na base do CRCN de acordo com o MCC são: Otsu, K-means eKapur. Na base da BioImLab os 3 melhores são: K-means, Sauvola e Johannsen.
Figura 37 – Representatividade MCC. (a) segmentação ideal (b) imagem resultante do algoritmoSeleção iterativa - 86,54% (c) imagem resultante do algoritmo Bernsen - 59,6%
(d) imagem resultante do algoritmo Two peaks - 22,69%
Elaborada pela autora
A Figura 37 acima mostra a correspondência do coeficiente de correlação deMatthews e a qualidade da segmentação. A Figura 37b contém a imagem resultante do

Capítulo 6. Validação de experimentos (resultados) 73
algoritmo Seleção iterativa com um MCC de 86,54%. A Figura 37c mostra a imagemresultante do algoritmo de Bernsen com um MCC razoável de 59,6% e a Figura 37d umaimagem inviável resultante do agoritmo Two peaks 22,69%. Nos três casos mostradosna Figura 37 pôde-se observar coerência entre o valor dado pela métrica e a qualidadeda segmentação.
Pode-se observar durante essa análise que alguns algoritmos são mais sensíveisàs diferentes características das bases de imagens do que outros. Dentre os algoritmosmais afetados pela mudança de base está o de Johannsen, Sauvola e Otsu, nessaordem, com resultados bem opostos, especialmente para as métricas precisão e MCC,como mostra a Figura 38.
Figura 38 – Comparação da relação algoritmo, base e métricas
Elaborado pela autora
Com base nas análises realizadas, os melhores algoritmos de segmentaçãono contexto de cromossomos avaliados neste trabalho foram escolhidos utilizando asquatro métricas - MCC, precisão, sensibilidade e acurácia, seguindo essa ordem deprioridade, ou seja, os algoritmos que obtiveram alta acurácia porém baixo coeficientede correlação de Matthews não foram classificados como bons, mas aqueles quealcançaram um MCC satisfatório e acurácia razoável (não necessariamente a melhorentre os algoritmos) foram selecionados como os melhores.
Dessa forma, os dois melhores algoritmos executados nas 35 imagens forne-cidas pelo CRCN foram: i) Otsu, com um MCC de 61,7% e precisão de 45,88% que,embora não sejam valores excelentes foi o algoritmo de maior MCC e segunda maiorprecisão dentre os algoritmos avaliados. Além disso, eles obtiveram ótimos resultadosde sensibilidade (88,3%) e acurácia (97,39%). ii) K-means, com o segundo melhor MCC

Capítulo 6. Validação de experimentos (resultados) 74
(58,48%) e terceira melhor precisão (44,58%) além de boa sensibilidade (86,67%) eexcelente acurácia (95,63%). A Tabela 1 abaixo mostra a média e desvio padrão, entreparênteses, dos valores resultantes de cada imagem para cada algoritmo e métrica deavaliação utilizada, em ordem decrescente seguindo o critério citado acima.
Tabela 1 – Média (desvio padrão) dos resultados de cada algoritmo para cada métrica - baseCRCN
MCC Precisão Sensibilidade Acurácia
Otsu 61,7% (0,13) 45,8% (0,15) 88,3% (0,15) 97,3% (0,01)
K-means 58,4% (0,20) 44,5% (0,23) 86,6% (0,17) 95,6% (0,05)
Kapur 50,2% (0,29) 38,1% (0,23) 73,9% (0,40) 97,1% (0,01)
Seleção iterativa 44,2% (0,27) 48,3% (0,31) 47,4% (0,32) 97,7% (0,01)
Bernsen 43,7% (0,14) 31,9% (0,17) 71,5% (0,08) 93,9% (0,07)
Percentage of black 39,9% (0,05) 18,3% (0,05) 98,3% (0,02) 91,2% (0,00)
Two peaks 39% (0,26) 30,5% (0,30) 75,1% (0,35) 88,2% (0,13)
Mean gray level 28,6% (0,24) 38,7% (0,33) 25,3% (0,28) 97,6% (0,01)
Sahoo 22,4% (0,26) 28,7% (0,21) 13,9% (0,29) 97,6% (0,01)
Niblack 16,6% (0,01) 5,2% (0,01) 90,7% (0,05) 67,4% (0,02)
Pun 14,1% (0,02) 3,9% (0,01) 99,9% (0,001) 51,8% (0,03)
Sauvola 10,7% (0,01) 3,3% (0,00) 91,8% (0,04) 48,2% (0,01)
Riddler 9,5% (0,17) 21,2% (0,29) 7,7% (0,19) 97,6% (0,01)
White 2,1% (0,04) 18,2% (0,31) 0,4% (0,01) 97,8% (0,007)
Johannsen 1,2% (0,07) 11,9% (0,31) 2,5% (0,15) 97,8% (0,01)
Elaborada pela autora
Já na base de imagens do BioImLab, os resultados foram um pouco diferentesprovavelmente por causa das diferentes características existente entre as bases de ima-gens como contraste, iluminação e excesso de ruídos. O algoritmo que obteve melhorresultado foi K-means com um MCC de 78,84% e precisão de 69,09% com excelentesensibilidade (96,6%) e acurácia (96,7%). O segundo melhor algoritmo foi o de Riddler,que embora tenha obtido o quarto melhor MCC (65,98%), obteve precisão (71,8%) eacurácia (96,25%) bem melhores do que os algoritmos que têm o segundo (Sauvola) eterceiro (Johansen) maior MCC, além de sensibilidade razoavelmente boa de 66,9%.Os resultados obtidos, na base BioImLab, foram colocados na Tabela 2, em ordemdecrescente de melhor algoritmo.

Capítulo 6. Validação de experimentos (resultados) 75
Tabela 2 – Média (desvio padrão) dos resultados de cada algoritmo para cada métrica - baseBioImLab
MCC Precisão Sensibilidade Acurácia
K-means 78,8% (0,15) 69% (0,25) 96,6% (0,07) 96,7% (0,03)
Riddler 65,9% (0,16) 71,8% (0,09) 66,9% (0,09) 96,2% (0,03)
Sauvola 68,4% (0,12) 57,4% (0,17) 88,1% (0,04) 95,6% (0,03)
Johannsen 67,5% (0,16) 50,5% (0,21) 99,9% (0,004) 93,6% (0,05)
Kapur 65,7% (0,27) 53,4% (0,26) 93,4% (0,22) 87,9% (0,25)
Two peaks 62,8% (0,30) 64,9% (0,34) 72% (0,38) 96% (0,20)
Sahoo 50,3% (0,20) 67,8% (0,32) 42% (0,12) 95,6% (0,02)
Seleção iterativa 41,2% (0,19) 65,4% (0,35) 30,6% (0,13) 95,2% (0,02)
Otsu 38,9% (0,28) 26% (0,22) 100% (0,36) 60,6% (0,36)
Mean gray level 36,1% (0,18) 65% (0,35) 24,8% (0,12) 95% (0,02)
White 19,1% (0,23) 13,1% (0,22) 99,3% (0,01) 35% (0,25)
Pun 18% (0,25) 14,4% (0,27) 99,3% (0,02) 28,9% (0,25)
Percentage of black 16,1% (0,22) 11,6% (0,19) 100% (0,23) 27% (0,23)
Niblack 10,5% (0,03) 6,2% (0,02) 96,8% (0,02) 27,4% (0,06)
Bernsen 3,5% (0,05) 5,5% (0,02) 74,6% (0,05) 35,1% (0,08)
Elaborada pela autora
Pôde-se observar nesta etapa que os algoritmos de agrupamento (clustering)obtiveram a melhor peformance visto que o algoritmo de Otsu e K-means, nessa ordem,foram escolhidos como os melhores para a base do CRCN e o K-means e Riddler osmelhores na base BioImLab. Contudo, é importante ressaltar que apesar do algoritmode K-means ter sido considerado bom em ambas as bases, o tempo de execução delefoi alto em comparação ao tempo dos demais algoritmos, especialmente para imagensgrandes como as da base de imagens do CRCN (2048x1536). A tabela 3 abaixo, mostrao tempo de execução, em segundos, para uma imagem de cada algoritmo para cadabase de imagens.

Capítulo 6. Validação de experimentos (resultados) 76
Tabela 3 – Tempo de execução (em segundos), para uma imagem, de cada algoritmo em cadabase de imagens
CRCN BioImLab
Otsu 0,195 0,054
Riddler 0,094 0,022
K-means 628,46 85,425
Seleção iterativa 0,328 0,125
Kapur 0,203 0,109
Pun 0,547 0,086
Johannsen 0,542 0,079
Sahoo 0,923 0,172
Two peaks 0,868 0,219
Bernsen 1,27 0,2575
Niblack 3,95 1,093
Sauvola 1,609 0,328
White 1,15 0,188
Mean gray level 0,797 0,079
Percentage of black 0,852 0,109
Elaborada pela autora
Note na tabela 3, que o tempo de execução do algoritmo de K-means foi bemsuperior aos demais. Para uma imagem, o algoritmo levou 628,46 segundos e 85,425segundos nas bases CRCN e BioImLab, respectivamente. Para 35 imagens, da basede imagens CRCN, seria necessário 6 horas e 6 minutos, aproximadamente. Para 500imagens, um tempo estimado de 3 dias, 15 horas e 15 minutos. Na base de imagensdo BioImLab, que possui resolução de 768x576, a execução de 35 imagens tem umtempo estimado de 49 minutos e para 500 imagens um tempo aproximado de 11 horase 51 minutos. Isso mostra o impacto da resolução da imagem no tempo de execuçãodos algoritmos.
6.3 Pós processamento
É importante ressaltar que os resultados mostrados nas Tabelas 1 e 2 foramobtidos da segmentação direta das imagens, isto é, não houve pré processamento comoetapa para remoção de ruídos ou melhora da qualidade das imagens o que de certaforma explica o fato da base do CRCN possuir valores obtidos nas métricas, em geral

Capítulo 6. Validação de experimentos (resultados) 77
mais baixos que o da BioImLab, pois na base da BioImLab há menor presença de ruídose variação de luminosidade. Dessa forma, para melhorar a qualidade da segmentação,e remover o ruídos que permaneceram, ou surgiram durante segmentação, foi utilizadoum pós processamento utilizando o filtro gaussiano com janela de tamanho 3x3 e atécnica morfológica de abertura com elemento estruturante de tamanho 5 e formaelíptica, obtendo um melhoramento mostrado no gráfico da Figura 39 abaixo.
Figura 39 – Gráfico comparativo entre segmentação de Otsu e Otsu + pós processamento
Elaborado pela autora
Na Figura 39, o gráfico mostra que houve uma pequena melhora na acurácia,precisão e MCC e uma redução na sensibilidade. Apesar de pequeno, o aumentoteve impacto na etapa posterior (contagem), devido à redução de ruídos. Na baseobtida do BioImLab não foi necessário a utilização de técnicas de pós processamento,pois o objetivo principal dessa etapa é a remoção dos pequenos ruídos, praticamenteinexistente nessa base. A Figura 40 mostra um exemplo do resultado/eficácia naaplicação dos filtros na etapa de pós processamento.

Capítulo 6. Validação de experimentos (resultados) 78
Figura 40 – Exemplo da imagem após etapa de segmentação (a) e depois do pósprocessamento (b)
Elaborada pela autora
6.4 Contagem
Após o pós processamento, a imagem foi submetida a uma espécie de identifi-cação de ruídos para eliminar os ruídos/objetos maiores que não foram removidos nopós processamento. A identificação baseada na área/tamanho dos objetos da imagemobteve um aumento nos valores da acurácia (1,5%), precisão (24,3%) e MCC (10,6%)em relação à segmentação obtida com o algoritmo de Otsu. Embora tenha diminuído asensibilidade em (10,7%), a qualidade da segmentação não foi impactada. A Figura 41mostra a influência das etapas nas métricas utilizadas na base do CRCN.
Figura 41 – Relação etapas x métricas - CRCN
Elaborado pela autora

Capítulo 6. Validação de experimentos (resultados) 79
Na base do BioImLab, o algoritmo de contagem foi aplicado diretamente nasimagens resultantes do algoritmo de K-means, classificado como melhor de acordocom as métricas. Ele obteve um melhoramento nos valores de todas as métricas comum aumento de 1,95% na acurácia, 0,03% na sensibilidade, 13,9% de precisão e 9,7%no MCC, como mostra o gráfico da Figura 42.
Figura 42 – Gráfico comparativo entre segmentação de k-means e k-means + contagem -BioImLab
Elaborado pela autora
Além das imagens, o algoritmo de contagem também retornou a quantidadede componentes conexos (teoricamente cromossomos, mas na prática ainda comalguns ruídos) de cada imagem, como explicado na Seção 5.5. Os resultados obtidosnão foram idênticos aos obtidos com as imagens rotuladas (segmentação ideal), masficaram bem próximos ao ideal. O algoritmo de contagem é parametrizado (limiteinferior da área dos componentes conexos e limite superior) por isso vários intervalosforam testados e o melhor parâmetro encontrado, para a base do CRCN foram oscomponentes conexos que possuem o valor de área compreendida num intervalo de150 a 6000 e do BioImLab um intervalo de 0 a 8000, ambos obtiveram a menor somadas diferenças. A Figura 43 mostra a semelhança entre o resultado ideal e o resultadoretornado pelo algoritmo, em cada base.

Capítulo 6. Validação de experimentos (resultados) 80
Figura 43 – Gráficos de dispersão dos valores resultantes e dos valores ideais (a) base CRCN(b) base BioImLab
Elaborado pela autora
Cada ponto dos gráficos acima representa uma imagem, e sua posição indica aquantidade de componentes conexos da imagem. Para as imagens rotuladas, os eixosx e y possuem o mesmo valor, esse valor indica a quantidade de componentes conexosda imagem. Nas imagens resultantes do algoritmo de contagem essa quantidade éindicada pelo eixo y. Os gráficos acima mostram que a base do BioImLab obteveuma quantidade de cromossomos (componentes conexos) mais próximos ao ideal(quantidade de componentes conexos contidos nas imagens rotuladas por especialistas)

Capítulo 6. Validação de experimentos (resultados) 81
embora não tenha usado técnicas de pós processamento o que comprova a influênciada forma de aquisição das imagens como mostra o artigo de Matta et al. (2013).
As Figuras 44 e 45 abaixo exemplificam a utilização de cada etapa de processa-mento utilizada neste trabalho, em cada base de imagem.
Figura 44 – Resultado das etapas CRCN (a) imagem original (b) imagem após binarização comalgoritmo de Otsu (c) imagem após pós processamento (d) após contagem
Elaborada pela autora

Capítulo 6. Validação de experimentos (resultados) 82
Figura 45 – Resultados de etapas BioImLab (a) imagem original (b) imagem binarizada comalgoritmo K-means (c) imagem após contagem
Elaborada pela autora

83
7 Conclusões e trabalhos futuros
Foi constatado que os algoritmos de agrupamento (clustering) obtiveram melhorperformance, sendo o algoritmo de Otsu e K-means os dois melhores na base doCRCN e K-means e Riddler os dois melhores na base do BioImLab. Observe que osmelhores algoritmos, em cada base, foram diferentes. Isso ocorreu, provavelmente,por causa das diferentes características existente entre as bases de imagens. Dessaforma, para selecionar o melhor algoritmo para uma determinada base de imagens decromossomos é necessário analisar as características das imagens quanto a presençade ruídos, iluminação e contraste. Embora o algoritmo de K-means tenha obtido bonsresultados em ambas as bases sua utilização pode se tornar inviável, em termosde tempo de execução, principalmente quando aplicado em imagens com grandesdimensões. Em geral, notou-se que a forma de aquisição das imagens tem grandeinfluência nos resultados dos algoritmos, mas alguns algoritmos como Johannsen,Sauvola e Otsu possuem maior sensibilidade às variações das bases de imagensapresentando resultados bem opostos em cada base.
Além da segmentação, foi implementado um algoritmo responsável pelo pósprocessamento e um algoritmo para identificação de ruídos e contagem. Os dois algorit-mos juntos conseguiram um aumento/melhoramento nos valores de precisão de 13,9%e 24,3% e no MCC de 9,7% e 10,6%. Os resultados referentes à contagem chegarampróximo à quantidade de cromossomos das imagens rotuladas manualmente, apresen-tando baixa dispersão. Isso indica que, se aplicado num futuro software, poderá auxiliaro profissional citogeneticista na realização da contagem. Devido à baixa presença deruídos da base de imagens BioImLab, ela obteve melhor similaridade, na etapa decontagem.
Com esse trabalho a primeira etapa na automatização do processo de dosimetriabiológica, segmentação, foi concluída. Porém, a parte referente a contagem pode sermelhorada com a utilização de técnicas mais sofisticadas na identificação dos ruídosque ainda permaneceram após o pós processamento. Como trabalhos futuros está aotimização do algoritmo de K-means com relação ao tempo de execução, o estudo detécnicas de detecção e separação de cromossomos colados e sobrepostos, a fim deobter uma contagem mais exata, além da classificação de cromossomos quanto aoseu tipo de aberração cromossômica bem como a quantificação dessas aberrações.

84
Referências
AINSBURY, E. A. et al. Review of Retrospective Dosimetry Techniques for ExternalIonising Radiation Exposures. v. 147, n. 4, p. 573 – 592, 2011. Citado na página 25.
BALDI, P. et al. Assessing the accuracy of prediction algorithms for classification: anoverview. Bioinformatics, v. 16, p. 412 – 424, 2000. Citado na página 55.
BALLARD, D. H.; BROWN, C. M. Computer Vision. In: . [S.l.]: Prentice- Hall, 1982.Citado na página 29.
BERTHOLDO, F. A. R. Técnicas de Limiarização para Melhorar a Qualidade Visualde Documentos Históricos. 2007. 75 p. Dissertação (Mestrado em Ciência daComputação) — Universidade Federal de Minas Gerais. Citado na página 32.
CRCN-NE. Pesquisa em Dosimetria (CRCN-NE). 1996. Disponível em: <http://www.crcn.gov.br/ptbr/pesquisa-em-dosimetria.php>. Acesso em: 12/10/2016. Citadona página 24.
CUNHA, K. de P. Aplicação de mapas auto organizáveis na classificação de aberraçõescromossômicas utilizando imagens de cromossomos humanos submetidos a radiaçãoionizante. 2015. Dissertação (Mestrado em Ciências na Área de Tecnologia Nuclear -\Reatores) — USP. Citado 2 vezes nas páginas 15 e 22.
DELGADO, J. U. RADIAÇÕES IONIZANTES E LEGISLAÇÃO PARA TRABALHADO-RES. 2000. Disponível em: <http://www.iaea.org/inis/collection/NCLCollectionStore/_Public/45/073/45073476.pdf>. Citado na página 24.
FACON, J. Técnicas de Processamento Digital de Imagens Aplicadas à Área da Saúde.In: . [S.l.: s.n.], 2013. cap. 3. Citado na página 37.
FACON, J. LIMIARIZAÇÃO POR ENTROPIA DE JOHANNSEN. 2016. Disponível em:<https://www.ppgia.pucpr.br/~facon/Binarizacao/LimiarEntropiaJohannsen3.PDF>.Citado na página 46.
FARIAS, A. M. L. de; LAURENCEL, L. da C. ESTAT´STICA DESCRITIVA. In: .[S.l.: s.n.], 2006. Citado na página 38.
FINNON, P.; LLOYD, D.; EDWARDS, A. An assessment of the metaphase findingcapability of the Cytoscan 110 . Mutation Research, Elsevier, p. 101 – 108, 1986.Citado na página 18.
GONZALEZ, R. C.; WOODS, R. E. Digital Image Processing. 3. ed. [S.l.]: Pearson,2007. Citado 4 vezes nas páginas 30, 31, 33 e 36.
GRISAN, E.; POLETTI, E.; RUGGERI, A. Automatic segmentation and disentangling ofchromosomes in q-band prometaphase images. IEEE, 2009. Citado na página 19.
IKEDA, D. T. Seleção Não-Supervisionada de Métodos de Binarização paraDocumentos Históricos. 2011. 50 p. Monografia (Bacharelado em Ciências daComputação) — Instituto de Matemática e Estatística Universidade de São Paulo.Citado 2 vezes nas páginas 50 e 51.

Referências 85
KHMELINSKII, A. Emparelhamento de cromossomas para propósitos de cariotipágem.2007. Dissertação (Mestrado em Engenharia Biomédica) — Universidade Técnica deLisboa. Citado na página 20.
KURTZ, G. C. Metodologias para detecção do centrômero no processo de identificaçãode cromossomos. 2011. Dissertação (Mestrado em Computação) — UniversidadeFederal de Santa Maria. Citado na página 20.
KURTZ, G. C. et al. Identificação automática de cromossomos humanos. XXI BrazilianSymposium on Computer Graphics and Image Processing, p. 33 – 36, 2008. Citado 2vezes nas páginas 17 e 19.
KURTZ, G. C. et al. Software para auxílio no processo de elaboração do cariótipo.2015. Citado 3 vezes nas páginas 16, 17 e 19.
LABORSUL. Disponível em: <http://www.laborsul.com.br/produtos/12>. Acesso em:22/11/2016. Citado na página 19.
M., I. M.; EMARY, E. On the application of artificial neural networks in analyzing andclassifying the human chromosomes. Journal of Computer Science, p. 72 – 75, 2006.ISSN 1549-3636. Citado na página 20.
MACQUEEN, J. SOME METHODS FOR CLASSIFICATION AND ANALYSIS OFMULTIVARIATE OBSERVATIONS. FIFTH BERKELEY SYMPOSIUM, v. 1, p. 281 – 297,1967. Citado na página 43.
MADIAN, N.; JAYANTHI, K.; DR.S.SURESH. Contour based segmentation ofchromosomes in g-band metaphase images. IEEE, 2015. Citado na página 19.
MARCOMINI, K. D.; SCHIABEL, H.; VERÇOSA, L. B. Aplicação de contornos ativospara a segmentação de nódulos em imagens de ultrassonografia da mama. RevistaBrasileira de Física Médica, 2012. Citado na página 54.
MATTA, M. C. D. Processamento de imagens em dosimetria citogenética. 2013.Dissertação (Mestrado em Dosimetria e Instrumentação Nuclear) — UFPE. Citado 4vezes nas páginas 18, 19, 24 e 25.
MATTA, M. C. D. et al. Processamento de imagens em Biodosimetria: Influência daqualidade das preparações cromossômicas. SCIENTIA PLENA, v. 9, n. 8, 2013.Citado 3 vezes nas páginas 17, 24 e 81.
MENDES, M. E. Verificação da taxa de alterações cromossômicas em sangue humanoirradiado em feixe gama. 2014. 91 p. Dissertação (Mestrado em Genética) — UFPE.Citado 5 vezes nas páginas 22, 24, 25, 26 e 56.
METAFER – Metafer Slide Scanning System. Disponível em: <http://www.metasystems-international.com/>. Acesso em: 22/11/2016. Citado na página 18.
MINAEE, S.; FOTOUHI, M.; KHALAJ, B. H. Geometric approach to fully automaticchromosome segmentation. 2014. Citado na página 19.
MÜLLER, D. N.; DARONCO, E. L. FILTROS ESPACIAIS PASSA-BAIXA. PROGRAMADE PÓS-GRADUAÇÃO EM COMPUTAÇÃO, Maio 2000. Citado na página 36.

Referências 86
NICKOLLS, E. et al. Preprocessing of images in an automated chromosome analysissystem. Pattern Recognition, v. 14, p. 219 – 229, 1980. Citado na página 17.
NUNES, F. L. S. Introdução ao Processamento de Imagens Médicas paraAuxílio ao Diagnóstico – Uma Visão Prática . 2006. Disponível em: <http://www.lbd.dcc.ufmg.br/colecoes/jai/2006/003.pdf>. Acesso em: 16/10/2016. Citado napágina 27.
NUSSBAUM; MCINNES; WILLARD. Thompson e Thompson: Genética Médica. 7aedition. ed. [S.l.]: Elsevier Editora Ltda, 2008. Citado 2 vezes nas páginas 15 e 16.
OTSU, N. A Threshold Selection Method from Gray-Level Histograms. IEEETRANSACTIONS ON SYSTEMS, MAN, AND CYBERNETICS, v. 9, n. 1, 1979. Citado2 vezes nas páginas 42 e 43.
PANTALEÃO, C. H. Z. Contribuição à Análise e Classificação Citogenética Baseadano Processamento Digital de Imagens e no Enfoque Lógico-Combinatório. 2003.Tese (Doutorado em Engenharia Elétrica) — Universidade Federal de Santa Catarina.Citado na página 17.
PARKER, J. Algorithms for Image Processing and Computer Vision. 2. ed. [S.l.]: WileyPublishing, Inc., 2011. Citado 4 vezes nas páginas 40, 44, 48 e 49.
POLETTI, E.; GRISAN, E.; RUGGERI, A. Automatic classification of chromosomes inq-band images. IEEE, Agosto 2008. Citado na página 20.
POLETTI, E. et al. A review of thresholding strategies applied to human chromosomesegmentation. Elsevier, 2012. Citado 4 vezes nas páginas 19, 44, 55 e 72.
QUEIROZ, J. E. R. de; GOMES, H. M. Introdução ao Processamento Digital deImagens. RITA(Revista de Informática Teórica e Aplicada), v. 8, n. 1, 2001. Citado 4vezes nas páginas 28, 30, 31 e 35.
RIDLER, T. W.; CALVARD, S. Picture Thresholding Using an Iterative Selection Method.IEEE TRANSACTIONS ON SYSTEMS, MAN, AND CYBERNETICS, v. 8, n. 8, August1978. Citado na página 43.
ROCHA, D. C. Mapas auto-organizável com campo receptivo adaptativo local parasegmentação de imagens. 2007. 117 p. Dissertação (Mestrado em Ciência daComputação) — UFPE. Disponível em: <http://repositorio.ufpe.br/xmlui/bitstream/handle/123456789/2704/arquivo6557_1.pdf?sequence=1&isAllowed=y>. Citado napágina 49.
SAHOO, P.; WILKINS, C.; YEAGER, J. THRESHOLD SELECTION USING RENYI’SENTROPY . Pattern Recognition, v. 30, n. 1, p. 73 – 84, Maio 1997. Citado na página48.
SAHOO, P. K. et al. A Survey of Thresholding Techniques. COMPUTER VISION,GRAPHICS, AND IMAGE PROCESSING, p. 233 – 260, 1981. Citado na página 45.
SANCHES, C. H. et al. Técnicas de Suavização de Imagens e Eliminação de Ruídos.In: Anais do EATI - Encontro Anual de Tecnologia da Informação e Semana Acadêmicade Tecnologia da Informação. [S.l.: s.n.], 2015. p. 21 – 30. Citado na página 36.

Referências 87
SEZGIN, M.; SANKUR, B. Survey over image thresholding techniques and quantitativeperformance evaluation. Journal of Electronic Imaging, p. 146 – 165, 2004. Citado 4vezes nas páginas 32, 41, 42 e 51.
SILVA, M. A. D. Dosimetria biológica em proteção radiológica: Elaboração de curvasrespostas para 60CO E137 CS. 1997. Dissertação (Programação de Pós- Graduaçãoem Tecnoçogia Nuclear) — IPEN/USP. Citado na página 25.
SINGH, O. I. et al. Local Contrast and Mean based Thresholding Technique in ImageBinarization. International Journal of Computer Applications, v. 51, n. 6, 2012. Citadona página 50.
SOUZA, L. P. Análise morfológica de imagens e classificação de aberraçõescromossômicas por meio de lógica fuzzy. 2011. 101 p. Dissertação (Mestrado emCiências na Área de Tecnologia Nuclear - Reatores) — USP. Citado 2 vezes naspáginas 17 e 22.
SRISANG, W.; JAROENSUTASINEE, K.; JAROENSUTASINEE, M. Segmentation ofoverlapping chromosome images using computational geometry. Walailak J Sci & Tech,2006. Citado 2 vezes nas páginas 17 e 19.
VERMA, R.; BABU, A. Human Chromosomes: Principles and Techniques. 2. ed. [S.l.]:McGraw-Hill, 1995. Citado 2 vezes nas páginas 15 e 16.

Apêndices

89
APÊNDICE A – Exemplos de imagens resultantes de cada algoritmo
avaliado na base de imagens fornecida pelo CRCN-NE.
Figura 46 – Imagem da base do CRCN e MCC correspondente. Segmentação ideal (a) Otsu -65,2% (b) Riddler - 64,2% (c) K-means - 70,9% (d) Seleção iterativa - 62,2% (e) Kapur -
70,4% (f) Pun - 11,6% (g) Jonhannsen - 0% (h) Sahoo - 63,3% (i) Two Peaks - 1% (j)Mean - 63,3% (k) Percentage of black - 35,5% (l) Bernsen - 60,3% (m) Niblack - 16,1%
(n) Sauvola - 9,5% (o) White - 4,6% (p)
Elaborada pela autora

90
APÊNDICE B – Exemplos de imagens resultantes de cada algoritmo
avaliado na base de imagens disponibilizada pelo BioImLab.
Figura 47 – Imagem da base do BioImLab e MCC correspondente. Segmentação ideal (a) Otsu -11% (b) Riddler - 79,6% (c) K-means - 88% (d) Seleção iterativa - 66,5% (e) Kapur -
89,3% (f) Pun - 11% (g) Jonhannsen - 99,9% (h) Sahoo - 74,6% (i) Two Peaks - 88% (j)Mean - 62,5% (k) Percentage of black - 11% (l) Bernsen - 16,5% (m) Niblack - 11,8%
(n) Sauvola - 42% (o) White - 12,7% (p)
Elaborada pela autora