UNIÃO DA TRANSCRIÇÃO E TRADUÇÃO DENTRO DO NÚCLEO DE CÉLULAS DE MAMÍFEROS
-
Upload
phallon-caddell -
Category
Documents
-
view
28 -
download
1
description
Transcript of UNIÃO DA TRANSCRIÇÃO E TRADUÇÃO DENTRO DO NÚCLEO DE CÉLULAS DE MAMÍFEROS
UNIÃO DA TRANSCRIÇÃO E TRADUÇÃO DENTRO DO NÚCLEO
DE CÉLULAS DE MAMÍFEROS
Coupled transcription and translation within nuclei of mammalian cells
Francisco J. Iborra, Dean A. Jackson, Peter R. Cook
Science vol 293, 1139-1142
Agosto 2001
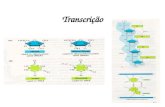
Introdução
Procariotos X Eucariotos
RNA polRNA pol
DNADNA
mRNA
mRNA
mRNA degradado
Tradução
Tradução
Ribossomo
Ribossomos Citoplasma
Citoplasma
Citoplasma
Transporte
Núcleo
Transcrição
Transcrição
mRNA maduro
Introdução
A membrana nuclear é a característica
que define os eucariotos.
Ela divide a célula em dois
compartimentos e assume-se que a tradução está
restrita somente a um deles - o citoplasma.
A idéia seria EVITAR a tradução antes
do splicing.
Introdução
Três evidências para que ocorra tradução
no núcleo:
1. Núcleo contém componentes necessários, mas
estes podem não estar ativos;
Introdução
2. Experimentos “pouco” controlados levam a
acreditar que núcleos podem aminoacilar tRNAs e
incorporar aminoácidos radioativos em proteínas.
Suspeita de contaminação
Núcleo Citoplasma
ProteínasMigração
Introdução
3. Degradação dos transcritos que possuem stop
códons prematuros (fenômeno conhecido como
“nonsense-mediated decay” - NMD).
UAA
Stop códon prematuro
Degradação
mRNA
mRNA
Introdução
Como pode a única maquinaria capaz
de reconhecer um stop códon - um ribossomo
citoplasmático ativo - disparar a degradação de
um transcrito que ainda está no núcleo?
Metodologia antiga de localização de sítios de tradução no núcleo:
[ 3H ] leucina tRNAleu - [ 3H ] leucina. Proteína
Desvantagem: tempo de incubação longo para a conversão da [ 3H ] leucina o que dá tempo à proteína recém sintetizada de entrar no núcleo.
Metodologia Antiga
Conversão
Síntese
Metodologia Atual
Um sistema de tradução in vitro que
utilizasse temperatura baixa e concentrações
subótimas de precursores, de modo que poucas
proteínas, que continham em média 350 resíduos
de aminoácidos, fossem completadas durante a
reação e estivessem aptas a escapar dos sítios de
síntese.
Metodologia Atual
Materiais a serem utilizados:
HCO3-
ATP
Na+
Mg2+
K+
[ 3H ] leu
19 aa
Aminoacil tRNA sintetases
GTP
Metodologia Atual
Condições do experimento:
Incubação a baixas temperaturas, 27.5o
Curto tempo, 3 minutos
Consequentemente, poucas proteínas seriam completadas durante a incubação.
Somente 15 resíduos seriam polimerizados.
Tamanho médio das proteínas seria de ~350aa.
Padronização do sistema
Verificação do efeito de inibidores na incorporação de [ 3H ] lisina durante a síntese de proteínas.
9%
Padronização do sistema
A mesma verificação foi feita ao se incorporar [35S] Met durante a síntese protéica.
Produtos marcados tinham o tamanho esperado de uma população de polipeptídeos recém-sintetizados normais.
Padronização do sistema
Para garantir, verificaram que a incorporação de [3H] Leu na presença de biotina-lisina-tRNA não inibia a síntese de proteínas.
Além disso, viram que a biotina era melhor imunolocalizador que os precursores radioativos.
Imunolocalização da biotina
A imunolocalização de polipeptídeos recém-
sintetizados com biotina é complicada pela existência
de biotina endógena.
Esta é encontrada solúvel e/ou
covalentemente ligada a carboxilases na matriz
mitocondrial.
A biotina solúvel foi extraída utilizando-se
tRNAs carregados. E a biotina covalentemente ligada
foi imunomarcada.
Imunolocalização da biotina
Biotina nas mitocôndrias foi
prontamente detectada, mas a dos
núcleos não.
Núcleo e citoplasma mais
marcados após incubação com
biotina-lisina-tRNA. A marcação
nuclear está concentrada em sítios
discretos.
Controle com cicloheximida
reduziu a marcação porque inibe a
síntese protéica.
Alguns peptídeos com biotina poderiam ter sido feitos no citoplasma e importados para dentro do núcleo, mas marcação similar foi obtida com núcleos isolados desprovidos de > 95% dos ribossomos citoplasmáticos.
Imunolocalização da biotina
Célula X Núcleo
Imunolocalização da biotina
Nenhum sinal mitocondrial foi visto do lado de fora dos núcleos, confirmando que a maior parte do citoplasma havia sido removida.
A remoção de > 95% dos ribossomos citoplasmáticos não afetou a intensidade nuclear.
Imunolocalização da biotina
A periferia nuclear não foi marcada,
por isso, a síntese de proteínas na membrana
nuclear externa seguida por importação não
foi a responsável pela marcação interna.
A mesma intensidade nucleoplasmática
foi obtida em amostras de células e núcleos
isolados.
Para provar que ribossomos citoplasmáticos não eram os responsáveis pela marcação do núcleo, foi utilizada a droga “thapsigargin” (bloqueador de importação nuclear).
Imunolocalização da biotina
Núcleo + thapsigargin
Proteínas
> 40KDa
Imunolocalização da biotina
Mesmo que os ribossomos citoplasmáticos tivessem entrado, eles não poderiam iniciar a tradução porque foi adicionado ATA (ácido aurintricarboxílico). Este inibe o início da tradução sem afetar a marcação do nucleoplasma.
Todos estes controles serviram para confirmar que a tradução citoplasmática não poderia ser considerada na marcação nuclear.
BODIPY-lisina-tRNA
Resultados similares foram obtidos com BODIPY-lisina -tRNA que também marca proteínas recém-sintetizadas em reações de tradução.
BODIPY-lisina -tRNA não tem o imprevisto da biotina, pois não é um composto endógeno.
O sinal nucleoplasmático constitui 14% do total.
A marcação citoplasmática foi cerca de 5 vezes mais forte que a nuclear.
BODIPY-lisina-tRNA
mRNA
mRNA
Núcleo
Citoplasma
A marcação com biotina-lisina-tRNA e
BODIPY-lisina-tRNA, que foi detectada com
métodos diferentes, deram resultados
similares.
Biotina X BODIPY-lisina-tRNA
Immunogold
Posteriormente, células permeabilizadas e
incubadas com biotina-lisina-tRNA foram
marcadas com “Immunogold” confirmando que o
interior nuclear continha cerca de 10% dos
peptídeos recém-sintetizados da célula.
Immunogold
Hipótese:
- Se toda a tradução ocorresse no citoplasma,
proteínas recém-sintetizadas entrariam e se
difundiriam por todo o núcleo; mas, se alguma
tradução estivesse unida à transcrição alguns
peptídeos nascentes teriam que ser encontrados
juntamente com transcritos nascentes.
Immunogold
Polipeptídeos recém-sintetizados e RNA foram estendidos na presença de biotina-lisina-tRNA e Br-UTP e também marcados com “Immunogold”. Os dois foram, muitas vezes, encontrados juntos.
Resultado final
Mostrou-se que :
- lisina ligada a três diferentes marcadores -
3H, biotina e BODIPY - é incorporada dentro de
sítios discretos no núcleo e que alguma desta
tradução nuclear depende de transcrição
simultânea pela RNA polimerase II.
Modelo
A partir deste resultado combinado ao resultado obtido no NMD propõe-se o seguinte modelo:
- Ribossomos são montados dentro do nucléolo e exportados para ambos nucleoplasma e citoplasma, onde eles se associarão com transcritos e se tornarão ativos;
- Exportação não precisa ser seletiva, talvez 10 vezes mais ribossomos terminem ativos no citoplasma, enquanto o nucleoplasma está junto à cromatina.
- Alguns ribossomos nucleares estão incorporados dentro dos sítios de transcrição do nucleoplasma que contêm muitas polimerases ativas e unidades de transcrição.
- Estes ribossomos “corrigem” transcritos recém-sintetizados enquanto eles emergem de polimerases.
Modelo
- Qualquer ribossomo acoplado detecta stop códons incorretamente posicionados, ativando a degradação destes transcritos; simultaneamente polipeptídeos truncados e indesejáveis são degradados por proteasomas.
- Se nenhum stop códon prematuro for encontrado, o transcrito deverá ser exportado para o citoplasma onde sofrerá múltiplas iniciações.
Modelo